 |
| サイトマップ | |
|
 |
| サイトマップ | |
|
Construct メニューには、コンストラクトの外観を変更するのに必要なあらゆるコントロールが用意されています。ある特定の特徴 (feature) の表示・非表示を切り替えたり、領域やマーク部位を追加することができます。コンストラクトウィンドウのグラフィックビューとシーケンスビューは Construct メニューで切り替えます。コンストラクトウィンドウにおけるコンストラクトの編集作業は基本的に Construct および Format メニューに用意されているツールを使って行います。
このメニューは、コンストラクトがグラフィック表示になっている場合はいつでも利用できます。
Construct > Magnification > Set Scale を使えば、コンストラクトの描画スケールを精密に設定することができます。このメニューを選択すると、図 8.28 に示すダイアログが表示されます。画面はその画面上の1インチ当たりのピクセル数で求められる解像度に制限されるので (通常は 72 ピクセル/インチ)、GCK には入力された値と共に画面上の実際のスケールが表示されます。Length of scale legend は、ウィンドウに描画されるスケールをあらわすバーの長さです。
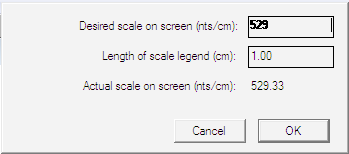 |
このダイアログでスケールを設定すると、コンストラクトを表示する具体的なスケールが GCK に定義されます。従って、コンストラクトウィンドウのサイズを変更しても、そのスケールは自動的には変更されません。ウィンドウを縮小すると、コンストラクトを移動するためのスクロールバーがアクティブになります。ウィンドウのサイズ変更と共にコンストラクトのサイズを自動的に変更できるようにするには、“Fit To Window” を選択してください。
Construct > Magnification > Zoom In (⇑-command-])/⇑-ctrl-]) を選択すると、挿入ポイント (または選択範囲) の位置を中心としてその表示が拡大されます。このコマンドを繰り返すたびにその表示は2倍に拡大されます。Construct > Magnification > Zoom Out (⇑-command-[/⇑-ctrl-[) を選択すると、操作を繰り返す度に半分に縮小されるので、コンストラクトの表示範囲を広げることができます。
Construct > Magnification > Fit To Window (⇑-command-W/⇑-ctrl-W) は、コンストラクトの表示比率をウィンドウにぴったり収まるサイズに変更します。Fit To Window を選択すると、ウィンドウのサイズが変更されるたびに、それに合わせてコンストラクトのスケールが変更され、再描画されます。このオプション以外を選択している場合は、ウィンドウサイズを変更しても、コンストラクトの再スケールは実行されません。コンストラクトを描画するスケールを特定の値に固定するには、“Set Scale” を選択します。
Construct > Magnification > Fit To Printer Page を選択すると、File > Page Setup… で定義した印刷用のページサイズいっぱいに埋まるようコンストラクトがリサイズされます。画面から切れた部分は、コンストラクトウィンドウの右側と下側に表示されるスクロールバーを使って確認することができます。再度全体を表示させたい場合は、Construct > Magnification > Fit to Window を選択します。
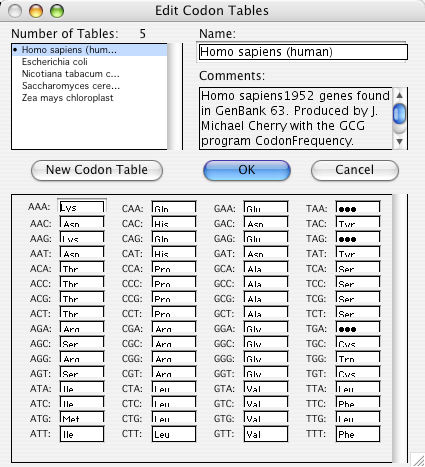
Construct > Features > Edit Codon Tables… を選択すると、図 8.29 に示すダイアログが表示されます。利用可能なコドン表の一覧は GCK Data フォルダで確認できます。図 8.29 でリストされている表は4つ (訳注:図では5つ) しかありませんa。このウィンドウの下半分は、現在選択している表で使用する全てのコドンとそれをあらわすアミノ酸の対応表です。リストで選択されたテーブル [この場合、Homo sapiens (human)] の左側には点が付けられ、Name ボックスにはその名称が表示され、この表に関連付けられたコメントがあれば、Comments ボックスにその内容が表示されます。OK ボタンを押すと、この表に加えられた変更内容が適用され、ウィンドウが閉じます。Cancel ボタンをクリックすると、変更内容を保存せずにウィンドウが閉じられます。New Codon Table を選択すると、現在表示されているコドン表の内容が複製され、新規名称で新しいテーブルを作成し、それの内容を編集することができます。新規作成したコドン表は、いずれも、必要があれば CCK のどこからでも自動的に利用できるようになります (例えば、“Find Open Reading Frames…” 参照)。新規テーブルが作成される場所は GCK Data フォルダとなります。
 |
| ※ a. Textco 社の塩基配列解析および分子生物学者のノートブックアプリケーション Gene Inspector® で作成したコドン表をお持ちであれば、それらを GCK Data フォルダに配置することで GCK で利用できるようになります。 |
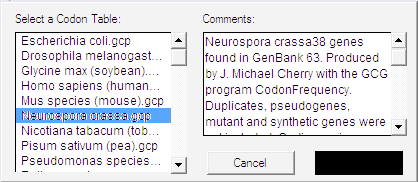
GCK で DNA 配列を翻訳したり、ORF を検索 (“Find Open Reading Frames…”) する場合、その翻訳にコドン表を使用します。使用するコドン表を標準テーブルにするには、ここで設定できます。図 8.30 に示すようなダイアログが表示されます。デフォルトで使用するコドン表を指定するには、左側のリストから目的のテーブルを選択し、OK ボタンをクリックします。Cancel をクリックすると、前に選択していたデフォルトの表を変更せずにダイアログを閉じます。提供されるテーブルにはそれに関連するコメントが含まれるものもあります。オリジナルのテーブルを追加したい場合は、そこにコメントを含めることができます。
 |
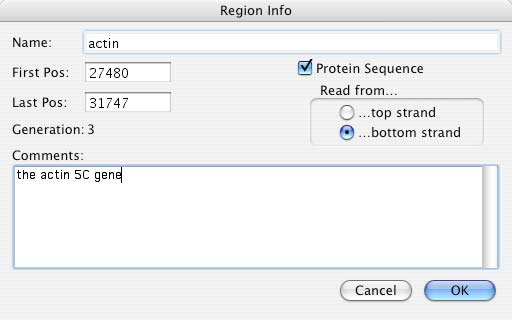
Construct > Features > Make Region (command-R/ctrl-R) を選択すると、選択したコンストラクトや塩基配列のセグメントに対応させる領域 (Region) を作成できます。DNA セグメントを何も選択していない場合は、図 8.31 に示すダイアログボックスに領域の端点 (First Pos と Last Pos) を入力する必要があります。いちど作成された領域については、それを選択状態にしたあと、Format メニューの適切なコマンドのいずれかを使用して変更を加えることができます。この領域の作成に使用するヌクレオチドの位置は、開始位置と終了位置を入力することで調整できます。ダイアログの Protein Sequence をクリックすると、コンストラクトをシーケンスで表示するときは、常に、デフォルトで指定したコドン表を使って (“Choose Codon Table…” 参照) GCK に塩基配列を翻訳させることができます。Protein Direction ボタン (訳注:Read from ボタン) を使えば、タンパク質配列の作成に使用する鎖を定義することができます。なお、選択範囲の向きは、Format > Lines メニューを使用して異なる矢印の向きを選択することで変更することもできます。
 |
Construct > Features > Find Open Reading Frames… メニュー項目は、図 8.32 に示すダイアログで設定したパラメータに基づいて、コンストラクトを検索して全ての読み枠 (Open reading frames) を見つけるものです。ORF 検索でコンストラクトの一部を選択した場合、調べる対象は、選択したセグメントのみとなります。DNA のセグメントが何も選択されていない場合は、コンストラクト全体が検索対象となります。ダイアログにはセグメントの開始位置 (Start offset) と終了位置 (End offset) が (ヌクレオチドの位置番号で)、翻訳に使用するコドン表と共に表示されます (コドン表の設定については “Choose Codon Table…” 参照)。ほとんどの場合は、標準テーブルで結構です。多くの教科書で使われている標準的な遺伝子コードに対応しているテーブルです。ミトコンドリアゲノムの一部や、その他の特殊な生物を取り扱っていれば、別なテーブルを選択する場合があるかもしれません。お使いの生物に対応するテーブルがない場合は、“Edit Codon Tables…” を使用して独自の表を作成することができます。
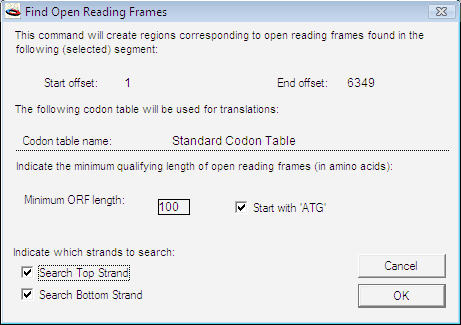 |
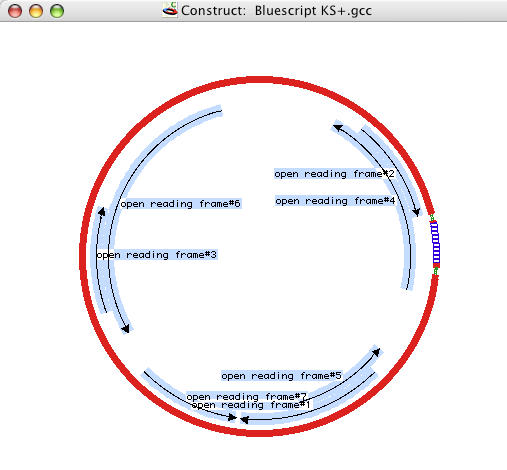
このダイアログボックスでは、レポートされる読み枠の最小の長さを指定する数値を入力することもできます。この長さに満たないものは表示されません。Start with ‘ATG’ チェックボックスを使えば、ATG で開始する読み枠のみを選択するか否かを指定できます。塩基配列が原核生物 (prokaryotic) の場合なら、このボックスにチェックを入れようと思うでしょう。真核生物 (eukaryotic) の塩基配列で下流エキソン (downstream exons) を探す場合は、このボックスにチェックを入れないように注意してください。最後に、検索する DNA 配列を上鎖 (Top Strand) と下鎖 (Bottom Strand) のいずれか一方又は両方にするかを指定することができます。読み枠 (ORF) が見つかると、自動的にコンストラクトウィンドウにマークと名称がつけられます。Show Region Names を選択していれば (“Show Region Names” 参照)、図 8.33 に示すように、その領域の横に名称が表示されます。
 |
ある DNA セグメントを選択状態にしたら、Construct > Feature > Define Intron を使用して、その部分をイントロンに定義することができます。イントロンにある塩基は、反転色で表示されます。通常の文字表示が赤であれば、反転表示は赤地に白となります。この DNA セグメントがコーディング領域の一部である場合、実際にタンパク質配列が作成される際にはこのヌクレオチドはスキップされます (イントロンと同じ)。これについては、Tutorial 15:翻訳におけるイントロンの読み飛ばし で詳しく説明してあります。イントロンを削除するには、イントロンにあるヌクレオチドを正確に選択したあと、Construct > Feature > Undefine Intron を選択します。イントロンの一部を含む領域を選択している場合、Construct > Feature > Expand Intron または Construct > Feature > Shrink Intron を使用して、イントロンの範囲を拡張したり縮めたりすることができます。
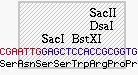
フレームについては、Tutorial 5:コンストラクト表示の修正 で既に説明してあります。フレームとは、コンストラクトの塩基配列リストの一部の周りに配置できるボックスです。Construct > Features > Make Frame を選択すると、選択した任意の塩基配列の周囲にフレームを作成することができます。図 8.34 に示すのはその一例です。一度作成したフレームは、チュートリアル5で説明したように Format メニューの項目を使って、その表示法を変更することができます。この図では、フレームがライトグレーに、塗潰しパターンがストライプに設定されています。この設定なら、塩基配列のリストが見にくくありません。
 |
フレームを選択するには以下の3つの方法があります:
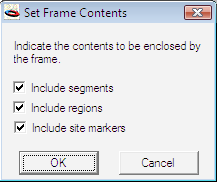
このトピックは、Tutorial 5:コンストラクト表示の修正 で既に説明してあります。図 8.34 に示すフレームには、塩基配列だけでなく、マーク部位と翻訳されたタンパク質配列も囲まれています。Construct > Features > Set Frame Contents を選択すると、フレームで実際に囲みたい内容を調整することができます。このオプションを選択すると、図 8.35 に示すダイアログが表示されます。このダイアログのチェックボックスを使用して、DNA セグメント、領域 (グラフィックの矢印または翻訳された配列) 、マーク部位からフレームに含める内容の組み合わせを指定することができます。
 |
これについては、Tutorial 2:サイトのマーキング で詳しく説明してあります。Construct > Features > Mark Sites (comment-M/ctrl-M) は、DNA 配列を検索し、選択したリストの各アイテムの認識配列と一致する位置にサイトマーカーを配置します。リストは、制限酵素や特定の配列を認識できるその他のアイテムが含まれた GCK ファイルです (Chapter 4: “リストウィンドウ” 参照)。Mark Sites を選択すると、図 8.36 に示すダイアログが表示されます。“部位のマーキング” で詳しく説明したとおり、このダイアログを使ってコンストラクトに部位をマークする方法を指定することができます。
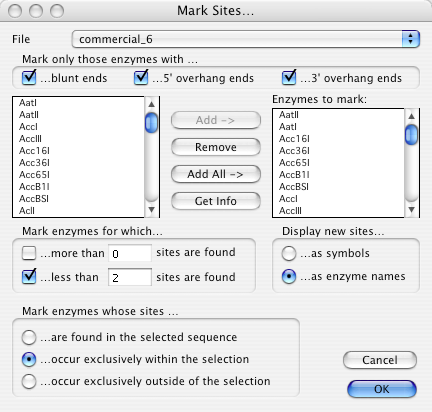 |
実行できるのは以下のとおりです:
それぞれの詳細は “部位のマーキング” をご覧ください。
Construct > Features > List Sites (command-L/ctrl-L) では、Construct > Features > Mark Sites (“Mark Sites…” 参照) と実行する操作が似ています。選択した制限酵素アイテムがリストファイルで指定された塩基配列と一致するコンストラクトの位置を検索します。Mark Sites では、コンストラクト上に実際にサイトマーカーを配置するのに対して、List Sites では、コンストラクト上の部位をあらわす一覧がテキストで作成されます。List Sites については、“部位 (Site) のリスト表示” で詳しい説明があります。
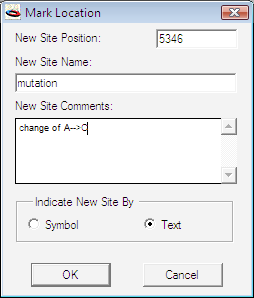
Construct > Features > Mark Location… は、コンストラクト上の指定した位置にマーカーを配置するものです。Mark Sites… とは異なり、Mark Location では、図 8.37 に示すダイアログボックスにサイトマーカーの正確な位置情報とその名称を入力する必要があります。マークを付けた位置に関連付けるコメントを入力することも可能です。入力されたコメントは、サイトマーカーをカット&ペーストしても情報は一緒に保持されます。リストウィンドウに格納するのが容易でない特徴 (feature) をあらわしたい場合に、位置のマーク付け (Mark Location) が役立ちます。例えば、転写開始部位 (transcription start sites)、ポリアデニル化信号 (polyadenylation signals)、スプライス部位 (splice sites) などに使用します。
 |
マーク位置 (Marked location) は、DNA をセグメントとして選択できるという点においてマーク部位 (marked site) と同様に機能します。GCK で用意された pBR322 コンストラクトの最も古い世代 (generation) では、ソースプラスミド pBR322 由来の3つの DNA 部分がセグメントとして定義されていますが、これはもともと特定のヌクレオチドにマーク位置を配置することによって定義されたものです。セグメントが定義されるとマーク位置は破棄されます。
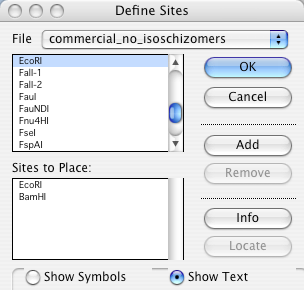
Construct > Features > Place Sites… (command-J/ctrl-J) は、Tutorial 11:汎用コンストラクトを使った作業 で既に説明してあります。Place Sites… を使えば、コンストラクトの特定の位置に認識配列を挿入することができます。この機能を使えば、塩基配列が不明であっても制限酵素部位を正確に配置した汎用コンストラクト (generic construct) を組み立てることができます。図 8.38 に示すダイアログボックスが表示されます。このダイアログは、コンストラクトに特定の部位を配置する最初の段階です。コンストラクトに配置させたい認識配列をもつ制限酵素を一番上のリストから選択して Add ボタンをクリックします (上のリストから制限酵素の名称をダブルクリックしても Sites to Place リストに表示させることも可能です)。図 8.38 では、AciI が選択され、下のリストに追加されています (訳注:現行の図 8.38 では EcoRI と BamHI が選択されている)。Sites to Place リストに追加されたアイテムを削除するには、そのアイテムを選択し、Remove ボタンをクリックします。
 |
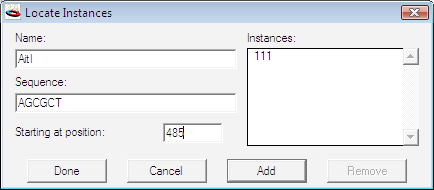
下のリストにアイテムが選択されると、Info ボタンと Locate ボタンが有効になります。Info をクリックすると、選択した制限酵素の切断部位とその他の関連情報をあらわす Get Info タイプのシンプルなダイアログボックスが編集不可能なウィンドウとして表示されます。Locate をクリックすると、図 8.39 に示すサブダイアログボックスが表示されます。この事例では制限酵素 AciI が選択されていますが、図に示すように、AciI (CCGC) の認識配列の配置をヌクレオチドの開始位置 100 と 485 に設定することにします (訳注:図が若干異なる)。Starting at position ボックスには2つめの開始位置 485 が入力されています。Add ボタンをクリックすると、ウィンドウ右側の Instances リストに 485 が追加されます。Instances リストに追加された項目を削除するには、削除したい項目を選択して Remove ボタンをクリックします。この制限酵素の位置をすべて定義したら Done ボタンをクリックします。メインダイアログ (図 8.38) に戻り、他の制限酵素の部位を配置する位置を定義することができます。
 |
なお、Place Sites では、コンストラクトのヌクレオチド配列に実際に変更が加わる点にご注意ください。指定した位置にあるコンストラクトの塩基配列は、選択した制限酵素認識配列をあらわす塩基配列に置き換わります。コンストラクトの N 記号だけではなく、ヌクレオチドが置き換わるのです!この操作は元に戻すことはできませんので、本当に実行すべきかどうか必ず確かめるようにしてください。
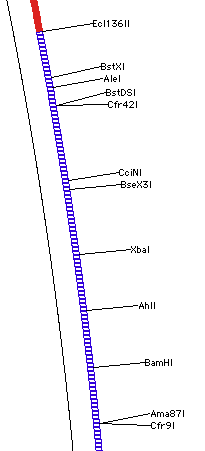
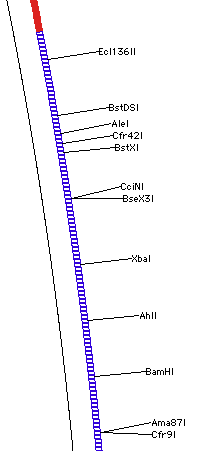
Construct > Features > Place site indicators at... は、サイトマーカーのコンストラクトにおける配置を指定するのに使用します。認識配列の開始点にマーカーを配置するか、実際の切断部位の左側に配置するかを選択するダイアログが表示されます。これは、制限酵素部位のマーカーの密集領域を高い拡大率でズームしたときしか目立った違いはあらわれません。図 8.40 に示すのは 3.6 nts/cm で描画したときの違いです。図 8.40 A は、認識配列にマーカーが配置されているものです。SmaI 部位がコンストラクトの DNA 配列と交わる位置に挿入ポイント (矢印で表示) が配置されていない点に注目してください (訳注:現行の図とは異なる説明)。SmaI マーカーテキストは SmaI 認識配列の開始点にありますが、挿入ポイント (新たに塩基配列がペーストされる場所) はその部位よりずっと下流にあります。図 8.40 B では、切断部位に SmaI マーカーテキストが配置されています。ここでは、挿入ポイントの位置に対応しています。伝統的には、制限酵素の名称の先頭文字がその制限酵素の認識配列の DNA 配列上の1文字目に来るように制限酵素の名称が配置されてきました。GCK では、いずれの方法も選択することができます。
| A | B |
 |
 |
コンストラクトを幾つも取り扱う場合、そこにある同一の特徴 (feature) にマークを付けたいことが良くあります。これらは一般に同一の塩基配列に基づいるので、そのセグメントをあらわすユニークなスタイルを作成することになります。例えば、お持ちのコンストラクトすべてについて、アンピシリン耐性遺伝子を、太さ 9 ピクセルの赤いセグメントで示そうと考えたとしましょう。また、プライマーや複製起点など特別に表示させたいその他の特徴もあるとしましょう。これらはいずれも、GCK の AutoFeatures を使用して実行することができます。これについては、Tutorial 21:AutoFeature の適用 で既に説明してあります。
塩基配列や、塩基配列の検索で許容するミスマッチの度合い、その特徴 (feature) のスタイルを AutoFeature 毎に定義することができます (セグメント又は領域に対して定義可能)。
Mark AutoFeatures を選択すると、GCK は選択した AutoFeature ファイルに定義された全てのアイテムを検索します。検索が終わったら、あらかじめ定義されたスタイルでその特徴にマークが付けられます。お望みならば、AutoFeatures でマークを付ける前に新しい世代で開始してもいいでしょう (“Start New Generation…” 参照)。
サイレント突然変異 (Silent mutations) は、DNA のコーディング情報には変更は加えずに DNA 配列を変更するものです。サイレント突然変異は、都合の良い制限酵素部位を作成したり、不都合な部位を削除するのによく利用されます。“Remove Site By Silent Mutation…” と Tutorial 6:DNA セグメントのクローニングとサイレント突然変異 参照。Construct > Features > Insert Sites By Silent Mutation を使えば、制限酵素部位を新たに配置したい場所に DNA セグメントを定義できます。これを開始するには、まずはじめに翻訳領域を選択する必要があります。どの読み枠を変換していいのか GCK には判断できませんので、この選択は重要です。図 8.41 に示すのがこれです。この事例では、7塩基認識部位の酵素のための新規制限酵素部位を挿入するのが目標です。領域を選択したら (図 8.41)、Insert Sites By Silent Mutations… を選択できるようになります。領域が選択されていなければ、このメニュー項目は選択できません。
 |
Insert Sites By Silent Mutations… を選択すると、図 8.42 に示すダイアログボックスが表示され、対象とする制限酵素を選択するよう尋ねられます。
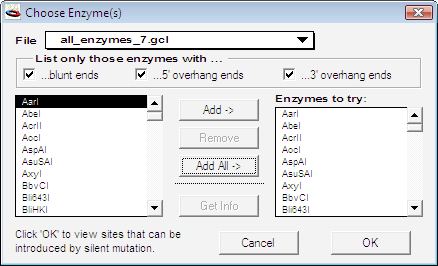 |
このダイアログボックスはいくつかのセクションに分けられています。一番上のポップアップリストでは、対象とする制限酵素として使用する制限酵素リストを選択することができます。図 8.42 は、all_enzymes_7.gcl を選択したものです。次のチェックボックスのかたまりからは、検索で考慮する切断部位の種類を選択することができます。左側の制限酵素リストには、現在選択されているリストファイルにあるすべての制限酵素が表示されます。左のリストからひとつまたは複数の制限酵素を選択して Add ボタンをクリックすると、それらは右側のリストに追加されます。右側のリストにある制限酵素はすべて、サイレント突然変異を挿入する対象となります。OK をクリックすると、DNA を調査するプロセスが開始し、突然変異によって変更される DNA の部位が特定され、DNA のコーディングには変更を加えない制限酵素部位が新たに作成されます。OK をクリックすると、図 8.43 に示すダイアログが表示されます。
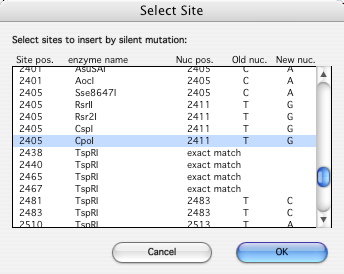 |
この図では、DNA の 2405 位置にある CpoI 部位が選択されています。この位置は CpoI の認識部位の開始点をあらわします。提案される変更内容は、ヌクレオチド 2411 の T を G に変更するものです。なお、この事例では CspI 部位と RsrII 部位も作成される点に注意してください。これは、T を G に変更すると、これら2つの制限酵素の認識部位も作成されるからです。仮に 2164 の SapI 部位を選択したとすれば、作成されるのはこの部位の1つだけとなります (2164 における G から A への変更は、対象となる制限酵素認識配列に対応するそれ以外の部位を生成しないからです)。
変更の加えられたヌクレオチドは、図 8.44 に示すように、他と区別しやすいように小文字に変換されます。
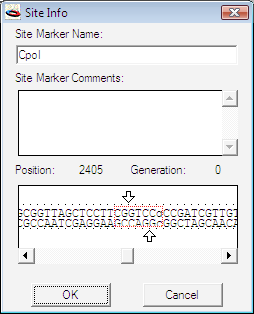 |
このダイアログボックスは、Construct > Get Info… を選択して表示される標準的な “Get Info…” ダイアログボックスです (“Get Info…” 参照)。なお、塩基配列の最後の g が小文字になっていますが、これは、オリジナルの塩基配列に変更が加えられたことをあらわしています。ボックスで囲まれた部分が CpoI 認識配列です。
Construct > Features > Remove Site By Silent Mutation… については、既に Tutorial 6:DNA セグメントのクローニングとサイレント突然変異 で説明してあります。このオプションは、単一点突然変異 (single point mutation) を作成し、その DNA セグメントのコーディングには変更を加えずに制限酵素認識部位を削除するものです。“Insert Sites By Silent Mutation…” と同じく、このメニュー項目を有効にするには、まずはじめに領域を選択する必要があります。
Display サブメニューには、コンストラクトのシーケンスビューおよびグラフィクスビューの両方に関連するオプションが用意されています。
Construct > Display > Show as Double/Single Stranded (⇑-command-D/⇑-ctrl-D) は、コンストラクトをシーケンスビューで表示する場合にのみ関係するオプションです。メニューの名称からもお分かりのとおり、このオプションを使えば、塩基配列の表示を二本鎖または一本鎖に切り替えることができます。選択とカット/コピー/ペースト操作は一本鎖モードと二本鎖モードで同様に機能します。二本鎖モードでは両方の鎖がハイライトで表示される点が異なります。
シーケンスビューのときだけ選択できるもうひとつのメニューは、Construct > Display > Show/Hide Positions (⇑-command-K/⇑-ctrl-K) です。これを選択すると、塩基配列の先頭にその位置番号が表示されます。表示されるのは、DNA 配列のある行の先頭のヌクレオチドの位置です。ペプチドを表示させている場合は、その行のアミノ酸の左端の位置になります。
コンストラクトが塩基配列で表示されていれば、Construct > Display > Show/Hide Line Borders (⇑-command-E/⇑-ctrl-E) を選択することができます。Line borders は、ドキュメントの印刷ページが複数行にまたがるような大きい場合に表示されるページ境界 (page border) と似たような働きをします (“Show/Hide Page Breaks” 参照)。ラインボーダーは塩基配列の一行毎に描画されます。ほとんどの場合、これを表示させる必要はありませんが、DNA が二本鎖で表示されており、かつ、領域とマーク部位の両方が表示されている場合、マーク部位と領域のセットが DNA 配列のどの行に関連づいているものなのか紛らわしくなる場合に使います。
Construct > Display > Set Line Spacing… メニュー項目を選択すると、塩基配列の上下の行間の最小値をピクセル単位で指定することができます。デフォルトで指定された値はゼロで、塩基配列行の行間は1文字分と等しくなります。この値をゼロから正の数に変更すると、塩基配列の各行は、その分だけ離れて移動します。行間 (Line Spacing) はデフォルトのプリファレンスで設定することができます。
このメニュー項目の対は、コンストラクトがシーケンスで表示されている場合のみ選択できます。Construct > Display > One Letter Amino Acid Code を選択すると、翻訳されたすべての塩基配列が1文字アミノ酸コードで表示されます。Construct > Display > Three Letter Amino Acid Code を選択すると、翻訳されたすべての塩基配列が3文字アミノ酸コードで表示されます。これはグローバルなセッティングであるので、変更内容はすべてのタンパク質配列に反映されます。同一コンストラクトにある異なる翻訳箇所を異なる文字数のアミノ酸コードで表示させることはできません。
コンストラクトをグラフィック表示している場合、Construct > Display > Show/Hide Scale Legend を選択できます。Show Scale Legend を選択すると、コンストラクトウィンドウに現在描画されているコンストラクトのスケールをあらわす水平線が図 8.45 のように表示されます。拡大率を “Fit to Window” に設定している状態でウィンドウサイズを変更すると、変更後のウィンドウのスケールにあわせてスケール凡例が更新されます。
 |
スケール凡例をダブルクリックすると、コンストラクトを描画したいスケールに設定することができます。これによって表示されるダイアログボックスは図 8.28 に示すようなものです。このダイアログボックスを使って、nucleotides/cm 単位のスケールとスケールインジケーターの長さを定義することができます。作成されるスケールは設定した値にできるだけ近い大きさになります。指定できるのはスクリーン上の1インチ当たりの固定ピクセル数なので、スクリーンを指定したスケールと厳密に一致させることは一般的に不可能です。しかし、Actual Scale on Screen の値を使えば、そのスケールを正確に定義できます。このダイアログボックスを使ってスケールを設定すると、このスケールの値に従ってコンストラクトが描画されます。従って、このスケールはウィンドウのサイズを変更しても変わりません。ウィンドウのサイズ変更に合わせてコンストラクトのサイズも変更させるようにするには、Construct > Magnification > Fit To Window を選択します (“Fit To Window” 参照) 。
コンストラクトタイトルは、環状コンストラクトの場合はその中央に、線状コンストラクトの場合ならその下に表示されるテキストです。このテキストには、一行目にファイルのタイトルが、二行目にコンストラクトの長さが表示されます。いずれのテキストも選択状態にしたあと Format メニューをでその属性を変更することができます。Construct > Display > Show Construct Title と Hide Construct Title を交互に選択することで、タイトルの表示と非表示を切り替えることができます。
コンストラクトタイトルは、マウスで選択してウィンドウの任意の場所に移動させることができますが、このタイトルを元の位置に戻したい場合があります。Construct > Display > Center Construct Title を選択すると、そのタイトルを環状コンストラクトの場合にはその中央に、線状コンストラクトの場合にはその下中央に戻ります。
この名称からお分かりのとおり、このメニュー項目は領域 (Region) の表示と非表示を切り替えるものです。この設定はグローバル設定なので、全ての領域が表示または非表示に変わります。領域 (region) とは、DNA の実際の部分ではなく、DNA に関する付加的情報をあらわす特徴 (feature) です (“関心領域と DNA の翻訳” 参照)。領域のひとつだけにこの属性を設定することはできません。状況に応じて表示をさせる領域を変更したい場合、また、どの領域も削除したくない場合には、クロノグラフィー機能を使ってこれを実現させることができます (“クロノグラフィ:コンストラクトの履歴の再現” 参照)。クロノグラフィの世代ごとに、表示させたい各領域セットを定義することができます。
この名称からお分かりのとおり、このメニュー項目はフレームの表示と非表示を切り替えるものです。この設定はグローバル設定なので、全てのフレームが表示または非表示に変わります。フレームとは、DNA 配列の表示上の装飾をあらわす特徴 (feature) です (“Make Frame…” および Tutorial 5:コンストラクト表示の修正 参照)。フレームのひとつだけにこの属性を設定することはできません。状況に応じて表示をさせるフレームを変更したい場合、また、どのフレームも削除したくない場合には、クロノグラフィー機能を使ってこれを実現させることができます (“クロノグラフィ:コンストラクトの履歴の再現” 参照)。クロノグラフィの世代ごとに、表示させたい各フレームセットを定義することができます。
この名称からお分かりのとおり、このメニュー項目はサイトマーカーの表示と非表示を切り替えるものです。この設定はグローバル設定なので、全てのサイトマーカーが表示または非表示に変わります。サイトマーカーとは、DNA の実際の部分ではなく、DNA に関する付加的情報をあらわす特徴 (feature) です (“Marking Sites” および Tutorial 2:サイトのマーキング 参照)。サイトマーカーのひとつだけにこの属性を設定することはできません。状況に応じて表示をさせるサイトマーカーを変更したい場合、また、どのサイトマーカーも削除したくない場合には、クロノグラフィー機能を使ってこれを実現させることができます (“クロノグラフィ:コンストラクトの履歴の再現” 参照)。クロノグラフィの世代ごとに、表示させたい各サイトマーカーのセットを定義することができます。
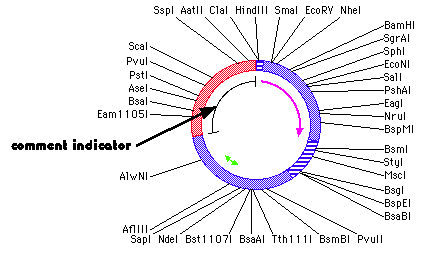
この名称からお分かりのとおり、このメニュー項目はコメントインジケーターの表示と非表示を切り替えるものです。コメントを添付しておけば、任意の DNA セグメントに注釈を格納し、いつでも簡単に表示させることができます。しかし、添付したコメントがどこにあるかわからず、目的のコメントがDNA のどのセグメントに格納されているか思い出せなくなる場合があるかも知れません。図 8.46 に示すのがコメントインジケーターで、これについては、“コンストラクトウィンドウ内のオブジェクトに対するコメントの添付” で既に説明してあります。コメントインジケーターの表示されている場所には、その DNA の特定セグメントにコメントが関連付けられていることを示します。セグメントインジケーターを一回クリックしてそれを選択状態にして、Construct > Get Info… を選択することで、そこに関連付けられているコメントを表示させることができます。コメントインジケーターをダブルクリックして、対応する DNA セグメントを選択状態にしたあと、Construct > Get Info… を選択することでそのコメントを表示させることもできます。コメントインジケーターは、操作上の観点からいえば、領域ととても似ていますが、コメントインジケーターと領域はそれぞれ個別に表示と非表示を切り替えることができる点、領域にはコメントが必須でない点、および、領域ではタンパク質配列を表示できる点が異なります。
 |
この名称からお分かりのとおり、このメニュー項目は世代 (generation) インジケーターの表示と非表示を切り替えるものです。世代 (generation) とは、GCK のクロノグラフィー機能の一部です。クロノグラフィについては、“クロノグラフィ:コンストラクトの履歴の再現” と Tutorial 7:クロノグラフィ (クローニング履歴の再現) で詳しく説明してあります。ひとつのコンストラクトに複数の世代が表示されている場合、セグメントにどの世代が表示されているのかをあらわすインジケーターを表示させることができます。セグメントごとに異なる世代を表示させることができるのでb、どの世代が現在表示されているのか把握するのが困難になる場合があります。この問題を解決するために、GCK では Construct > Display > Show/Hide Generations を使って、それぞれの世代の違いをあらわすことができるようになっています。世代表示を有効にすると、コンストラクトの外側に現在表示されている世代では表示されていない全てのセグメントに、図 3.35 のようなラインが追加されます。現在の世代で表示されているセグメントには、それ以外にインジケーターラインはありません。直前の世代で表示される断片は、環状コンストラクトの外側にグレーのインジケーターで表示されます。
| ※ b. セグメントを選択すると、Chronography メニューを使って任意の世代を表示させることができます。この方法を使ってセグメントごとに異なる世代で表示させることができます。 |
任意のコンストラクトは、塩基配列をグラフィックで表示することも、塩基配列としてリスト表示することもできます。いずれを選択しても、その表示法が異なるだけで、いずれも表示している内容は同じです。Construct > Display > Display Sequence と Construct > Display > Display Graphic (shift-command-/shift-ctrl-/) を選択すると、コンストラクトの2つの表示法を切り替えることができます。
コンストラクトには、いつでも汎用ヌクレオチド文字列 (N) を挿入することができます。Construct メニューの Construct > Insert Ns… (command-H/ctrl-H) を選択すると、図 8.47 に示すダイアログボックスが表示されます。たとえコンストラクトウィンドウが空の場合であっても、制限酵素地図を作成したり、塩基配列がまだわかっていないコンストラクトを形作るのに役立ちます。Place Sites オプション (“Place Sites…” 参照) と組み合わせて使えば、コンストラクトのサイズと制限酵素部位の幾つかの場所しか分かっていない場合であっても、完全な図を作成することができます。
 |
Construct > Insert Typing… は、コンストラクトをシーケンスビューで表示しているときしか選択できません。メニューからこれを選択するか、カーソルを塩基配列の任意の場所に置いて入力を開始することによって使用することができます。これについては、“キーボードを使った新規ヌクレオチドの挿入” で説明してあります。
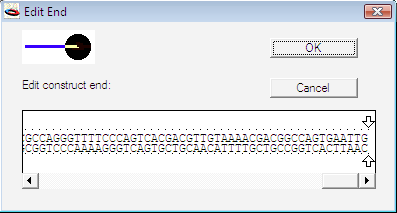
線状コンストラクトの末端にカーソルを置くと、Construct > Edit Construct End… (command-E/ctrl-E) メニューオプションを選択できます。このメニュー項目を選択すると、図 8.48 に示すようなダイアログボックスが表示されます。ダイアログの左上の図に示されているように、表示されている塩基配列は、コンストラクトの右端を表しています。この末端は、Gene Construction Kit の標準的なやり方で編集することができます。矢印をドラッグしてヌクレオチドを鎖から削ったり、別な場所でマウスをクリックして矢印を移動できます。
 |
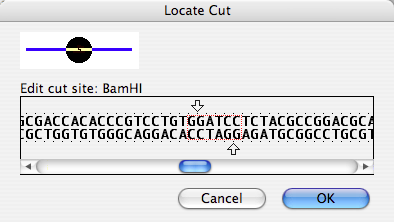
カーソルがコンストラクトの末端の一方ではなくマーク部位にある場合、メニューは Edit Cut Site… に変わり、図 8.49 に示すようなダイアログボックスが表示されます。矢印をドラッグすることによって、特定の制限酵素の切断部位を調整することができます。切断部位に変更を加えた場合は、制限酵素の名称を必ず変更するようにしてください。
 |
Construct > Find Sequence… (command-F/ctrl-F) については、既に “塩基配列の検索” で説明してあります。DNA の標準文字を使用すれば (“Legal Nucleotide Characters” 参照)、コンストラクト全体を検索し、検索配列に一致する箇所をハイライトで表示させることができます。検索は、カーソルの現在位置から開始し、時計回り (環状 DNA の場合) または、5’ --> 3’ 方向 (線状 DNA の場合) に実行されます。線状 DNA の場合は、末端にたどりつくと検索は停止します。環状コンストラクトの場合は、検索を継続させることができます。
コンストラクトをグラフィカルビューで表示している場合、コンストラクトの形状が環状 (circular) か線状 (linear) かによって、Construct > Redefine Origin または Construct > Set Beginning Position (command-1/ctrl-1) を選択することができます。環状コンストラクトの場合に、Redefine Origin を選択すると、挿入ポイントの現在の位置がコンストラクトの新しい複製起点になるようにコンストラクトの配置を変更することができます。これによって、画面上のコンストラクトの複製起点はコンストラクトの一番上に回転します。線状コンストラクトの場合に、Set Beginning Position を選択すると、図 8.50 に示すダイアログを使用して、塩基配列の開始ヌクレオチド番号を設定しなおすことができます。例えば、作業中の DNA に非常に多数の塩基配列からなるセグメントがあり、そのセグメントが DNA の先頭のセグメントでない場合に役立ちます。このような場合、セグメントの番号付けで、そのヌクレオチドが実際に存在する塩基配列全体の位置 (例えば 86 など) から開始すれば一貫性を保つのが簡単になります。また、翻訳の開始位置を相対的に示す場合もあるでしょう (-266 など)。論文用の図を作成する場合に、この機能は非常に重宝します。
 |
フレームは、塩基配列の特定の部分をハイライトで表示させるために使用します。フレームについては、Tutorial 5:コンストラクト表示の修正 で既に説明してあります。コンストラクトのシーケンスビューを使えば、フレームはもちろんサイトマーカー、DNA セグメント、および領域を選択することができます。フレームをどのように選択したらよいか迷う場合があるかもしれません。Construct > Select Frame を選択することで、カーソルの形状が "クロスヘアー" (十字) に変わりますので、この状態でフレームの内部をクリックすると、そのフレームを選択状態にすることができます。フレーム内部の DNA 配列をトリプルクリックするか、オプションキーを押しながら選択したいフレームの内部をクリックすることで、目的のフレームを選択することもできます。フレームが選択状態になったら、Format メニューの項目を利用して、フレームの外観を変更することができます。
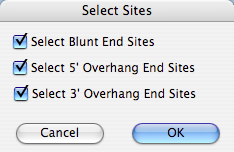
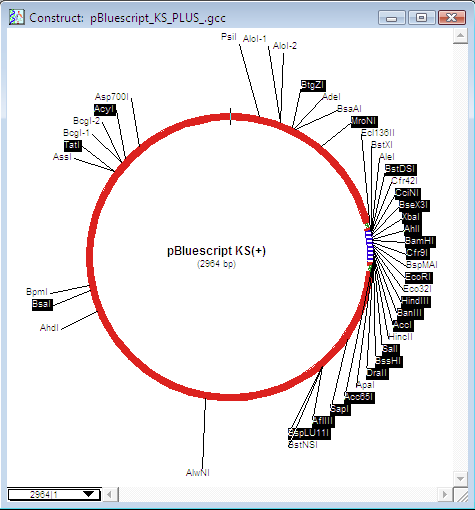
Construct > Select Sites By Kind を選択すると、制限酵素認識部位の属性に応じて任意のコンストラクトのマーク部位の一部だけを選択することができます。図 8.51 に示すダイアログボックスを使って、選択したい部位の種類を指定します。すべてのユニークな部位を表示した pBR322 についてこの選択を実行したのが図 8.52 になります。5' 突出末端と 3' 突出末端の部位から区別したい場合、選択されたこれらすべてのカラーやフォントを変更する場合に便利です。5' と 3' 部位を選択して、識別カラーをコーディングすることも可能です。こうした情報が一目で見れるようになるので、クローニングストラテジーを設計するのが容易になります。
 |
 |
その名称が示すとおり、Go to Position を使えば、お持ちの塩基配列情報の指定した位置に移動することができます。位置情報をひとつ入力すると、GCK のカーソルがその位置まで移動します (挿入ポイントは指定された塩基配列の位置の直前に配置されます)。位置情報を 500-600 のようなヌクレオチドの範囲で入力すると、指定された塩基配列の位置まで移動したあと、500 から 600 のヌクレオチドがハイライトで表示されます。これについては、 Tutorial 19:Go To Position で説明してあります。
Get Info… (command-I/ctrl-I) を選択すると、コンストラクトウィンドウで選択された任意のオブジェクトに関する情報が表示されます。Get Info… で情報が表示される対象は、コンストラクトのセグメント、マーク部位、領域です。表示されるダイアログボックスはいずれも似ていますが、選択したオブジェクトによって表示される内容は異なります。この中のいくつかは既に説明してあります。図 3.8 は領域 (Region) に関する Get Info ボックスです。図 3.7 は部位 (site) に関する Get Info ボックスです。図 3.4 はセグメントに関する get Info ボックスです。なお、イランストレーションウィンドウでコンストラクトがターゲットになっている場合も Get Info… を利用することができます。
Construct > General Info… は、コンストラクト全体に関する情報が表示される点で Get Info… とは異なります。基本情報 (General information) はコンストラクトのどのオブジェクトとも関連はありません。図 3.6 に示すのはそのダイアログです。基本情報 (General information) は常にコンストラクトと共に関連づけられます。セグメントを別のウィンドウにカット&ペーストすると、その基本情報はペースト先のウィンドウ (ファイル) でも引き継がれ、失われることはありません。これに対して、セグメントのコメントはセグメントが切り離された時点で無くなります。コンストラクト全体をコピーし、新規ウィンドウにコピーすると基本情報がコピーされます。コンストラクト全体を別のコンストラクトの一部としてペーストすると、元になる基本情報はペーストによって作成される新規セグメントの情報になります。
Construct > Search Comments… オプションは、“コメントの検索” で既に説明してあります。全ての世代のコンストラクトに関連付けられている全てのコメントを検索することができます。図 3.19 に示すように、直観的な方法で基本情報 (general info)、セグメント、領域、マーク部位、およびクロノグラフィ世代を検索することができます。条件に一致したものは一覧で表示され、それぞれのコメントを表示させることができます。
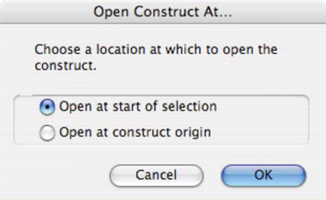
Construct > Make Linear/Make Circular (command-\/ctrl-\) はコンストラクトの表示を線状 (linear) または環状 (circular) に切り替えるものです。環状コンストラクトを線状コンストラクトにすると、コンストラクトの開き方について、挿入ポイントまたは複製起点 (origin) を選択するダイアログが図 8.53 のように表示されます。線状コンストラクトを環状コンストラクトに切り替える場合、線状コンストラクトの両端が複製起点になります。必要な場合は、ライゲーション (結合) ダイアログボックスが表示されます。
 |