 |
| サイトマップ | |
|
 |
| サイトマップ | |
|
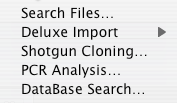 |
Tools > Search Files… については、既に Tutorial 8:コメントの検索とファイルの検索 で説明してあります。ファイル検索では、ディスク上の Gene Construction Kit ファイルからコメントとして入力されたキーワードや DNA 配列に一致するものを検索できます。このオプションの使用法に関する詳細な説明はチュートリアルをご覧ください。
お使いのコンストラクトに情報をまめに入力するのであれば、Search Files を様々なコンストラクトの保管場所を把握するのに使うことができます。例えば、すべてのコンストラクトに関連する共通のコメントとして、そのコンストラクトの保管場所を入力しておけば、ファイル検索を使って、例えばアクチン・コンストラクトは全て冷蔵庫番号2番にあることを見つけ出すことができるでしょう。GCK をコンストラクトの保管場所と塩基配列情報を提供するシンプルなデータベース管理システムとして利用できるわけです。
このメニューには6つのサブメニューがあります。GCK には、GenBank features table から各種アイテムを取り出し、GCK 固有の特徴 (feature) に変換する機能が装備されています。このような処理を実行するために用意されているのがこのサブメニューにあるメニュー項目です。Tutorial 13: Deluxe Import 機能を使用した GenBank シーケンスファイルの読み込みには、この処理に関する具体的な説明があります。
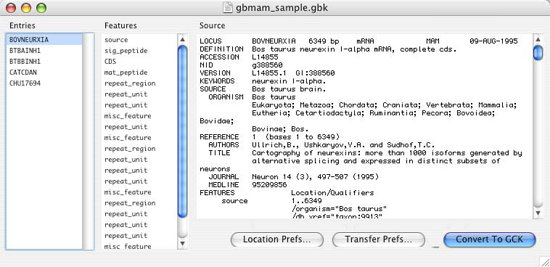
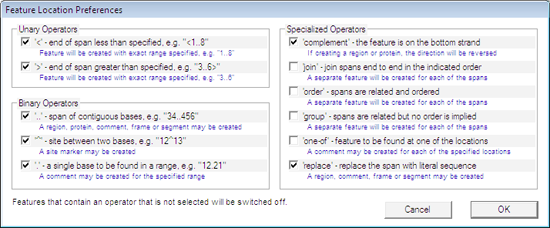
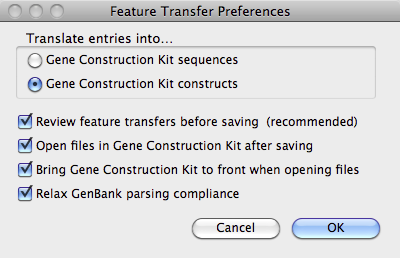
既に GenBank または EMBL ファイルをハードディスクにダウンロードしていれば、このオプションを選択して、それらを GCK ファイルに変換することができます。ファイルを開くと、図 8.22 に示すダイアログが表示されます。このファイルの場合には、5種類の塩基配列が含まれており、その中の1番目が現在選択されています。Location Prefs ボタンを使うと、GenBank ファイルで指定された位置 (Location) を GCK のどの位置に変換するか定義できます。これを選択すると図 8.23 に示すダイアログが表示されます。このダイアログを使用して、実際に使用するオプションを選択し、それらをどのように解釈するかを指定することができます。Transfer Prefs ボタンを押すと図 8.24 に示すダイアログが表示されます。すべてのパラメータの設定が完了したら、その内容は GCK に記憶されるので、同じ操作を繰り返す必要はありません。ファイルのインポートには図 2.67 で説明したように Conversion Defaults が使用されます。
 |
 |
 |
Retrieve Sequence メニューオプションを使えば、GenBank または EMBL データベースから特定の塩基配列を検索 (Retrieve) することができます。図 8.25 に示すようなダイアログに、検索する塩基配列の accession number または keyword を入力します。
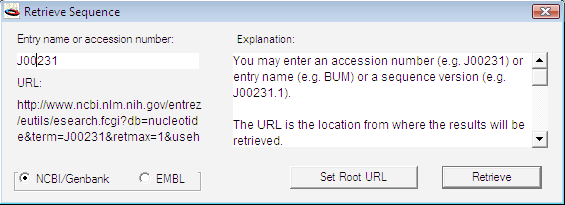 |
ローカルネットワークに GenBank フォーマットでセットアップされたデータベースがあれば、Set Root URL ボタンをクリックして、そのデータベースを選択することができます。プライベートなデータベースを検索する場合に役立ちます。なお、この検索を実行させるには、ターゲットデータベースが GenBank または EMBL データベースと同じフォーマットである必要があります。
このオプションについては、Tutorial 14: GenBank を使った検索とシーケンスファイルの取り込み で既に説明してあります。キーワードを任意数のカテゴリに入力して、GenBank 検索を実行できます。カテゴリは、図 2.71 に示すようなポップアップメニューを使って選択できます。
Conversion Defaults は、GenBank table の各種 feature を GCK の feature にどのように変換するかを定義するものです。Tutorial 13: Deluxe Import 機能を使用した GenBank シーケンスファイルの読み込み で説明したように、GenBank の feature を GCK コンストラクトとして最終的にあらわす方法を何通りも選択することができます。図 2.67 に示すのは、Conversion Defaults ウィンドウです。Conversion Defaults で指定する個々の対応は、実行する変換をそれぞれ定義するのに使用します。ただし、変換を実行する際は、デフォルトの変換を個別に選択するか、すべて選択するかのどちらかを選ぶことができます。
好みの Conversion Defaults セットの定義が完了したら、それをファイルとして保存しておき、必要があれば後から呼び出して使うことができます。Conversion Defaults が複数あれば、このオプションを使って、それぞれ個別のファイルを作成することができます。
このメニュー項目を使えば、Save Conversion Defaults で保存した任意のファイルを開き、別なデフォルトセットとして使用することができます。
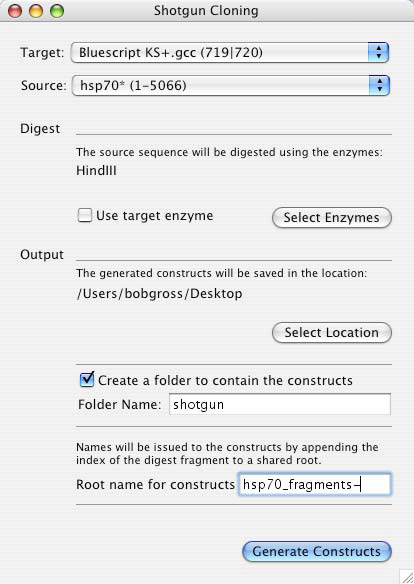
ショットガン・クローニングとは、DNA セグメントを断片化し、標的ベクターの制限酵素部位にそれらを配置するプロセスのことです。Gene Construction Kit では、任意の制限酵素を DNA の消化 (digest) に選択し、ベクター DNA の標的部位として使用することができます。従って、このプロセスでは2種類の DNA が関係することになります:ソース DNA とターゲット DNA です。出力される個々のファイルはもちろん、消化 (digest) で使用する DNA と制限酵素は、すべて Shotgun Cloning ウィンドウで選択します。
Target および Source ポップアップメニューには、既に開いているコンストラクトが表示されます。サイトマーカーを選択していれば、それらがデフォルトの制限酵素として利用されます。コンストラクトファイルを開いていなければ、ポップアップメニューから Browse ボタンを選択して使用したいコンストラクトを選択できます。ファイルを選択すると GCK は自動的にそれを開きます。Use Target Enzyme にチェックを入れると、ターゲット DNA で選択したサイトマーカーが、デフォルトの制限酵素に使用されます。チェックが入っていない場合は、Select Enzymes ボタンをクリックすることで任意の制限酵素を選択することができます。なお、選択する制限酵素の末端は、ターゲットの部位に適合する必要がある点に注意してください。適合しない末端は、ターゲット部位と結合 (ligate) しません。Select Location を選択すると、新たに作成されるファイルのホームとなるフォルダを選択 (フォルダを新規作成) できます。Folder Name フィールドには新規フォルダの名称を入力します。最後に、Root name for constructs フィールドで生成されるすべてのファイル名に使用するプレフィクス (接頭辞) を設定できます。生成されるファイルにはプレフィクスで指定したテキストで始まる連番がつけられます。Generate Constructs をクリックすると、プロセスは完了します。
 |
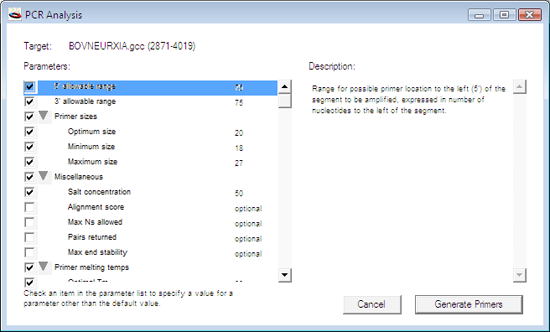
ポリメラーゼ連鎖反応 (PCR: Polymerase Chain Reaction) は、小規模な DNA 断片を増幅するための方法です。GCK は米ホワイトヘッド研究所 (Whitehead Institute) で開発されたコードを許可を得て使用しています。このコードは、Primer 3 と呼ばれるもので、PCR 解析コードの中でも高く評価されているもののひとつです。PCR 解析の具体的な使用法については、Tutorial 16: PCR 解析 をご覧ください。
PCR 解析を開始すると、図 8.27 に示すダイアログが表示されます。ウィンドウの左側にある Parameters リストには、ユーザーが定義できる多数のパラメータがあります。リスト内の任意の項目をクリックすると、ウィンドウの左側にある Description フィールドに項目の説明が表示されます。Generate Primers ボタンをクリックすると、GCK はプライマー対の候補となるリストを返します (図 2.81 参照)。プライマー対のいずれかを選択し、Show Info ボタンをクリックすると、そのプライマー対の様々な属性のスコアが詳細に表示されます。プライマー対のいずれかを選択して Add Primer ボタンをクリックすると、それをコンストラクトに追加できます。プライマー対の追加が完了したら、Done ボタンをクリックしてウィンドウを閉じます。コンストラクトに追加したプライマー対はいずれも、逆向きのインジケーターが対になって表示されます。ひとつはコンストラクト DNA の上鎖、もうひとつは下鎖です。プライマー対のいずれかをクリックして、Get Info... を選択すると、Show Info ウィンドウで選択できたのと同じ情報が表示されます。
 |
生物学者に関わりのある情報を備えたデータベースは、インターネット上に多数存在します。データベース検索機能の使用法については、Tutorial 18:データベース検索機能 で詳しく説明してあります。基本的な考え方は、キーワード、タンパク質または DNA の塩基配列を GCK に渡すと、プログラムがその情報をウェブ上の単一または複数のデータベースサイトに送信するというものです。
この検索は、Textco BioSoftware 社のウェブサーバーで稼働する DB サーチエンジンを通じて管理されます。DB サーバーは、DNA、タンパク質、または、キーワードを情報として受け取り、クエリーを各種ウェブサイトに送信します。DB サーバーには、クエリーをどのようにフォーマットするかという情報が含まれており、クエリー情報を各サイトにあわせて適切にフォーマットします。サイトのフォーマットの仕様に変更が加えられたら、DB サーバーも更新されることになっているので、個別のアプリケーションをアップデートする必要はありません。この仕組みは、GCK のクライアント側にアップデートを強いることなく、DB サーバーを新規サイトに更新し、古くなったサイトを削除できることも意味します。
新しいサイトに関するご提案が何かありましたら、info@textco.com (開発元の Textco Biosoftware 社) 宛までご連絡ください。