 |
| サイトマップ | |
|
 |
| サイトマップ | |
|
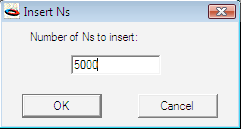
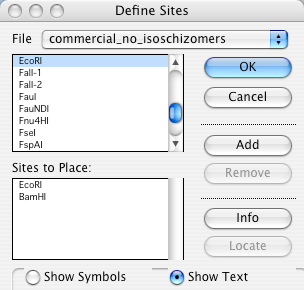
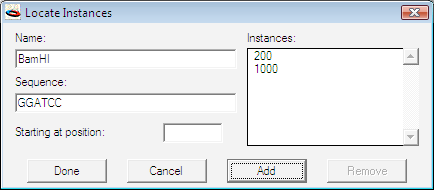
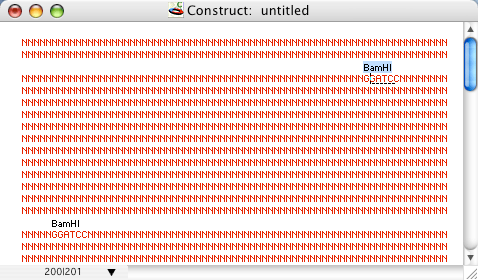
DNA の一部を使って作業に取り掛かろうとする場合、クローニングされた DNA 断片の制限酵素地図は分かっていても、その塩基配列が未知であることがよくあります。このような汎用コンストラクトを GCK で作成し、そのセグメントをあたかもその配列が既知の DNA の一部であるかのように操作することができます。
 |
 |
 |
 |
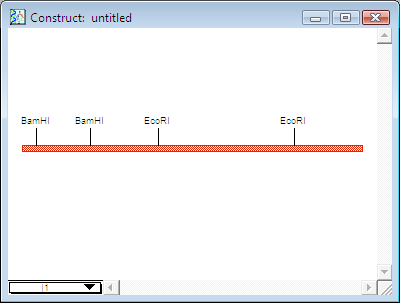 |
以上でこのチュートリアルは終了です。開いているウィンドウを (変更内容を保存せずに) 閉じてください。