バージョン 12 の新しい機能
仮想クローニング法の増加
CodonCode Aligner 12.0 は、既存の制限クローニングおよび Gibson Assembly シミュレータに加えて、TA クローニング、指向性 TOPO クローニング、平滑末端 PCR および TOPO クローニングを導入しています。
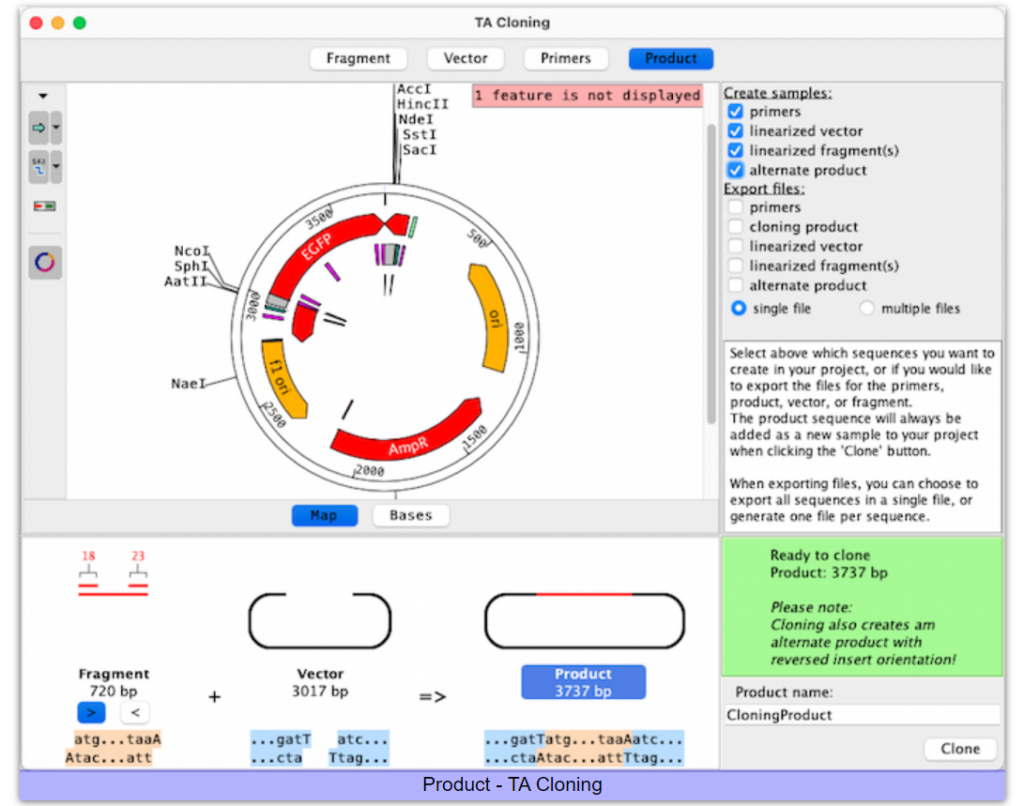
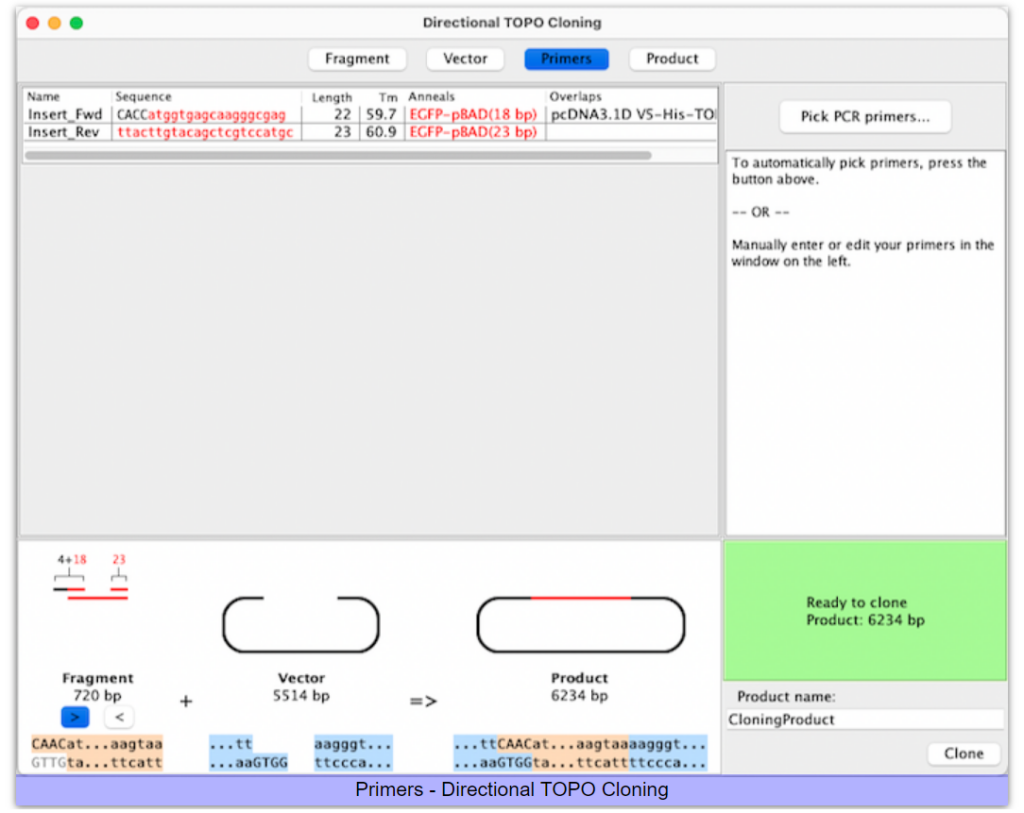
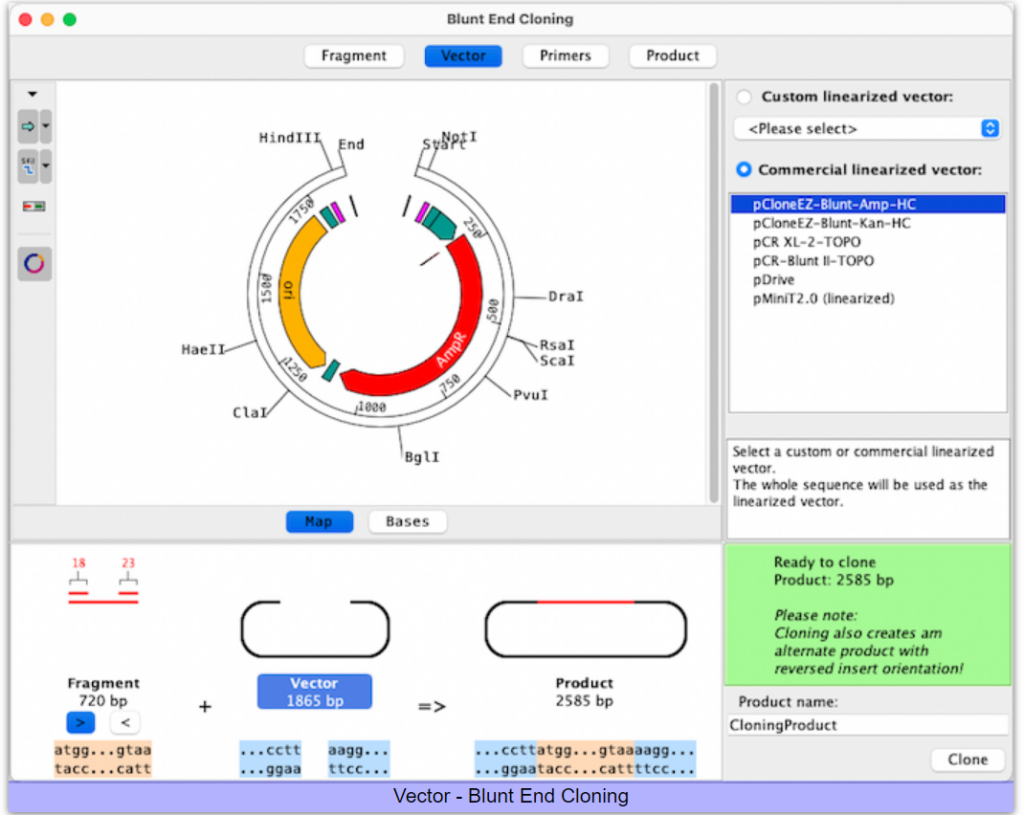
すべてのシーケンスは、フィーチャーとカットされたサイトを示すマップスタイルで表示することも、ベースレベルに表示することもできます。表示するカットサイトとフィーチャーは、クローニングビューの左側にあるツールバー ボタンを使用してカスタマイズできます。下部のオーバービューには、ベクター、フラグメント、プライマー、プロダクトがグラフィカルに表示されます。
すでに含まれている一般的なクローニングベクターから選択するか、独自のカスタムベクターを使用します。
Tm の設定に応じて最適なプライマーを自動的に選択します。また、必要に応じてスペーサーを追加したり、リーディングフレームを調整したりするなど、プライマーを手動で編集することもできます。
最後に、プライマーとフラグメントを直接エクスポートすることを選択し、クローニング実験からどのシーケンスを作成して CodonCode Aligner プロジェクトに追加すべきかを選択します。
フィーチャーの転送
サンプル間でのフィーチャー (タグ) の転送が、かつてないほど容易になりました。2 つのシーケンスを選択し、検索するフィーチャーの最小 % ID を選択するだけです。また、検索されたフィーチャーのどれを転送するかを手動で調整することもできます。
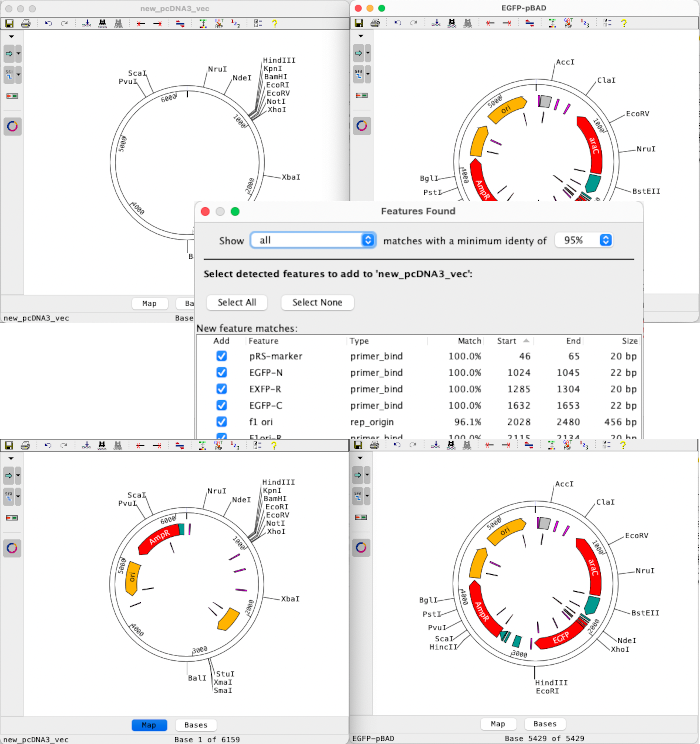
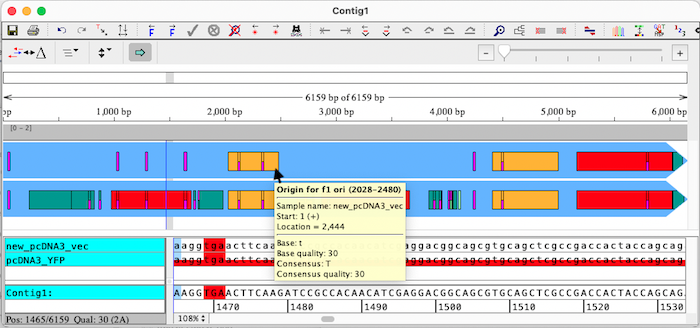
コンティグオーバービューのフィーチャーを参照
Aligner 12 では、コンティグオーバービューの矢印にフィーチャー (タグ) が表示されるようになりました。これにより、シーケンス間のフィーチャーを簡単に比較できます。マウスオーバーすると詳細なフィーチャー情報が表示され、矢印のサイズや表示するフィーチャーをカスタマイズできます。
CodonCode Aligner 12.0 は、Windows 11 および macOS Sequoia(15) と互換性があります。
(2024/12/03)
バージョン 11 の新しい機能
Gibson Assembly (ギブソンアセンブリ)
バーチャルギブソンアセンブリのサポートにより、最大 15 個のフラグメントでクローニング実験をシミュレートできます。
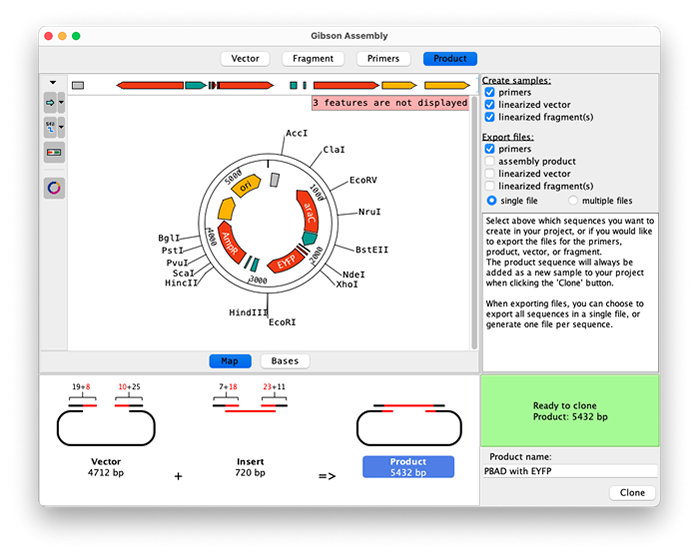
ベクター、フラグメント、プロダクトのシーケンスは、線形マップ、円形マップ、または塩基レベルで表示することができます。フィーチャー (注釈) をクリックする、既存の選択範囲を反転させる、酵素を選択する、塩基番号を入力する、単にShift-クリックを使用するなど、簡単なオプションのいずれかを使用してクローニングする領域を選択します。
ビューの左側にあるツールバーボタンには、表示するマップ、フィーチャー、酵素を変更するオプションが含まれています。
最大 15 個のフラグメントを追加でき、必要に応じて修正、順序付け、削除ができます。
Tm とオーバーラップの設定に応じて、最適なプライマーを自動的に選択します。また、必要に応じてスペーサーを追加したり、リーディングフレームを調整するなど、手動でプライマーを編集することもできます。
ベクター、フラグメント、プライマー、プロダクトの概要がグラフィカルに表示されます。
クローニング実験中に、プライマーとフラグメントを直接エクスポートし、CodonCode Aligner プロジェクトにどの配列を作成するかを選択します。
共通フィーチャーの検出とアノテーション
Aligner 11 では、プラスミドに共通するフィーチャーを検出し、アノテーションするオプションが追加されました。検出するフィーチャーの類似度を設定し、どのフィーチャーにアノテーションを付けるかを選択できます。
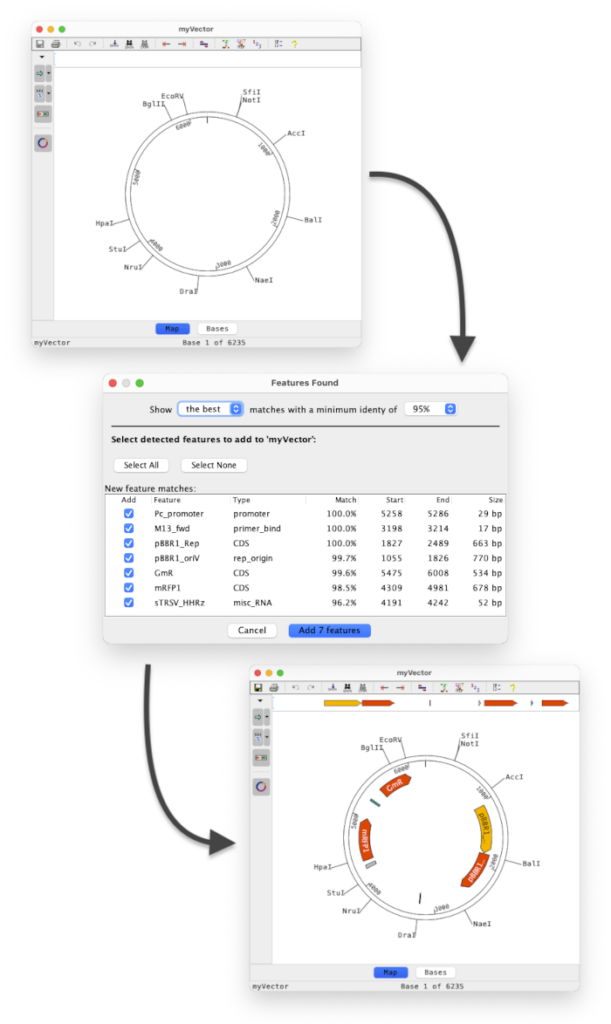
上の例では、95% 以上の同一性を持つ共通フィーチャーを検出してアノテーションする前と後のベクトルを示しています。
シーケンス開始位置の同期機能
円形シーケンスの開始位置を同期させる機能によって、類似したサンプルを並べて比較することが簡単にできるようになりました。以下は、類似点はあるが開始位置が異なる 2 つのベクトルを、開始位置を同期させる前と後で比較した例です。
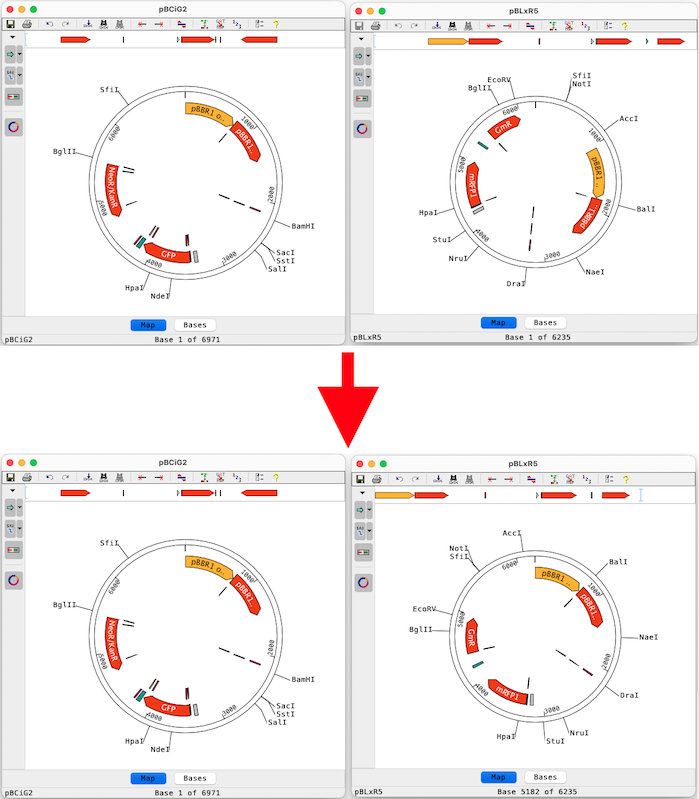
自動同期機能を使用する代わりに、手動で開始ベースを設定することも可能です。
その他
- GFF ファイルからフィーチャーをインポート
- 環状配列における開始位置の設定
- 環状配列のアラインメントを改善
- 最近使ったファイルのインポート
- Windows 11 および macOS Ventura (13) 対応
バージョン 10 の新しい機能
制限クローニング
CodonCode Aligner 10 では、仮想的な制限クローニングを実行できます。
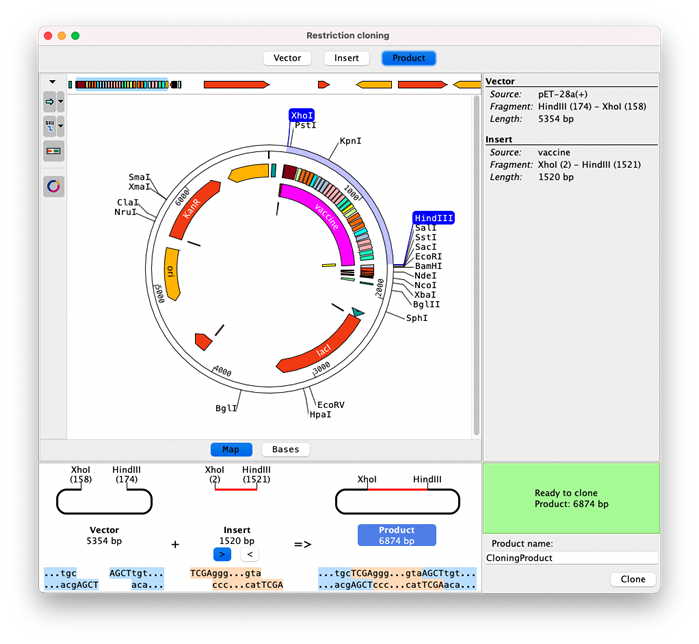
線形または円形のマップ、又は、塩基レベルとしてベクターと挿入シーケンスを表示できます。制限酵素やフィーチャー (注釈) をクリックすることで酵素とクローンするセクションを容易に選択できます。
画面下の部分には、ベクターと挿入シーケンスがどのように組み合わされているか、切断部位に基づいて生成物ができるかが表示されます。
画面左側にあるツールバーボタンには、どのマップ、フィーチャー、および、制限酵素を表示するかを変更するためのオプションが用意されています。例えば、一度切断した制限酵素だけを表示するという具合です。
シーケンスマップ
Aligner 10 には、クローニング機能と連携したシーケンスマップを表示できる機能が追加されました。円形と線形のマップを表示したら、そこにカスタマイズ可能なフィーチャーと酵素切断部位が表示されます。
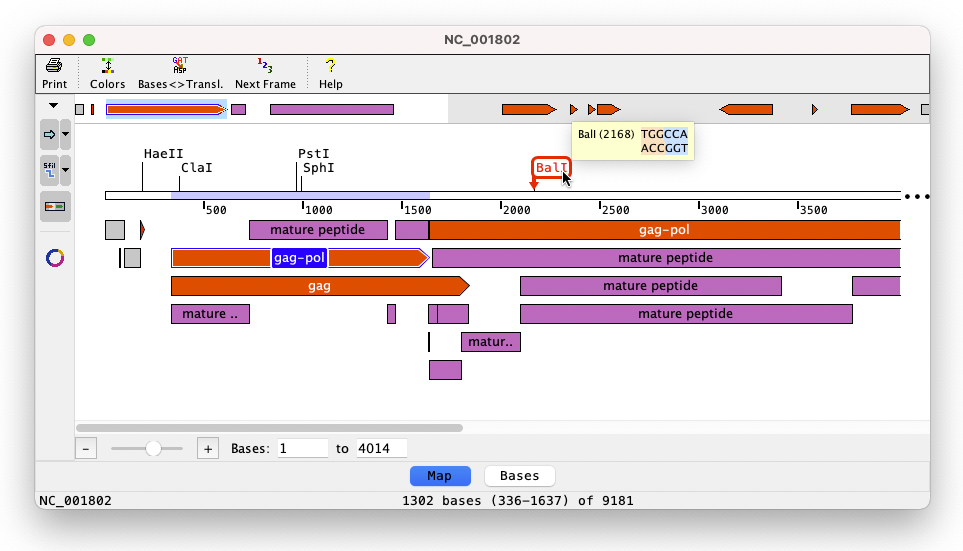
画面上部にミニマップとして概要が表示されます。シーケンス内の特定の領域を選択するには、そのフィーチャーをクリックするか、Shift キーをおしながら2つの酵素をクリックします。マウスオーバーすると、切断部位とフィーチャーに関する詳しい情報が表示されます。また、画面下のズームオプションを使用すれば、特定の領域をズームすることができます。特定の領域を選択したり移動したあとにマップ表示と塩基表示を素早く切り替えることができます。また、画面左にあるボタンを使えば、表示するフィーチャーや酵素といったマップオプションをカスタマイズできます。
対立遺伝子の取り出し
CodonCode Aligner の “Separate Alleles” は、小規模な次世代シーケンスプロジェクトにおいて二倍体生物由来の対立遺伝子間の連鎖を特定するための機能です。対をなす末端配列の中の連鎖する対立遺伝子をあらわした事例を以下に示します。
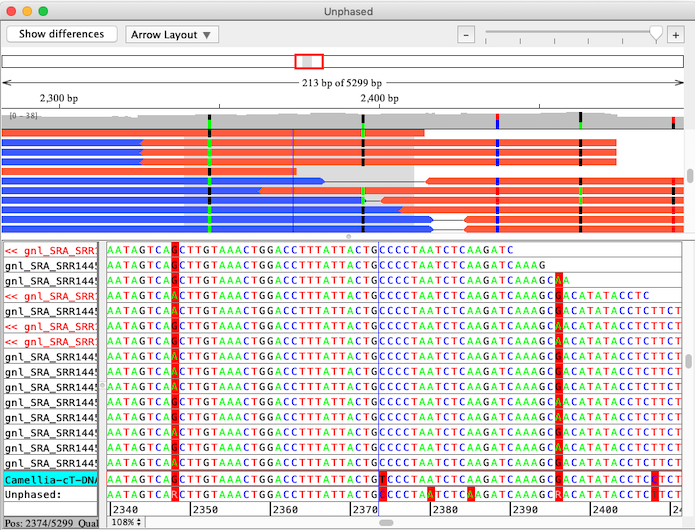
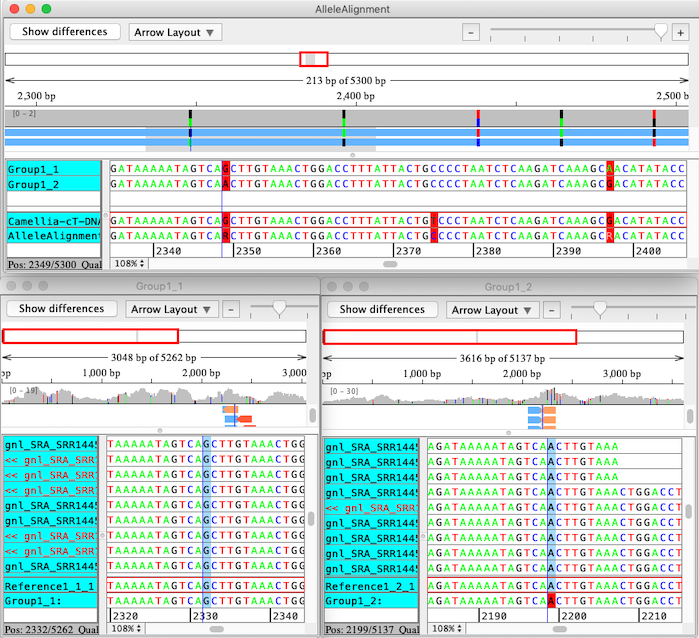
CodonCode Aligner は、複数ステップのプロセスを経て各配列を参照配列にアラインメントし、対立遺伝子の連鎖を識別し、配列対を連鎖グループとして取り出します。この分析の最終結果は、連鎖した対立遺伝子をグループに分けた個々のコンティグです。これらは、CodonCode Aligner の “Compare contigs” 機能を使ってオリジナルの参照配列にアラインメントされます。以下のスクリーンショットは、このような分析を行った結果を示します。画面上には、”contig of contigs” の対立遺伝子のアライメントが、画面下には、対立遺伝子に固有のコンティグが表示されています。
その他
- アミノ酸の一文字翻訳を塩基レベルで表示
- 配列内の開始コドンと終止コドンをハイライト表示
- カスタマイズ可能なグループ、又は、グループ分け無しで塩基を表示
バージョン 9 の新しい機能
Base (塩基) ビュー
CodonCode Aligner 9 には、機能と制限酵素切断部位を表示する新しい Base (塩基) ビューが導入されています。
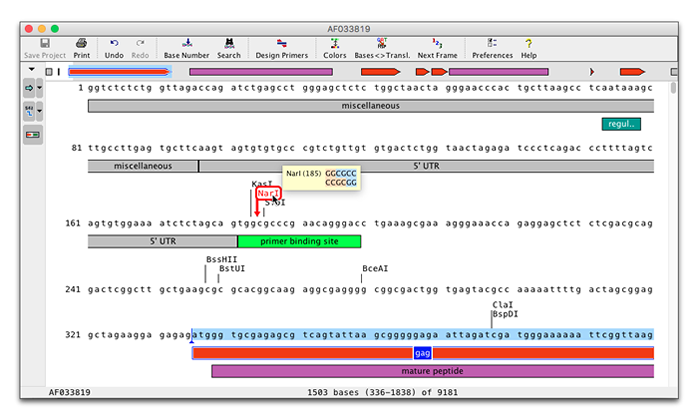
feature (特徴) をクリックすると、features とそれがカバーする塩基が選択されます。 Shift キーを押しながらクリックすると、選択範囲が広がります。 最初に制限酵素名をクリックし、2 番目の酵素名を Shift キーを押しながらクリックして、制限断片を選択できます。
マウスオーバーすると、feature と切断部位に関する情報が表示されます。 上部のミニマップは feature の概要を示し、シーケンス内のクイックナビゲーションを可能にします。
Base ビューの左側にあるツールバーボタンには、特定の feature と酵素の表示非表示、または選択するオプションが含まれます。たとえば、一度切断した酵素のみを表示します。
シーケンス選択
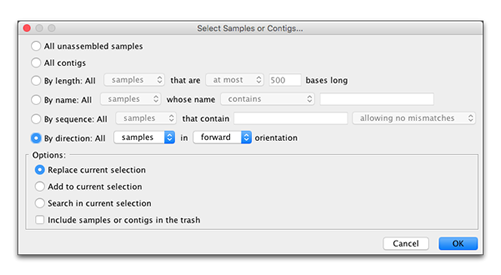
Aligner 9.0 では、長さ、名前、シーケンス、方向、およびアセンブリ状態に基づいてサンプルとコンティグを選択する機能が追加されています。
この機能は大きなプロジェクトで作業する場合に特に役立ち、スクロールしてシーケンスを選択する必要がなくなります。
Search in the current selection オプションを使用して、これらの選択方法を一度に複数使用できます。
EMBL および Genbank Feature のサポート
Aligner 9.0 に Genbank と EMBL 形式のシーケンスエクスポート機能が新しく追加されました。
また、すべての Genbank および EMBL feature を Aligner 9 にインポートして、ユーザー設定に基づいて表示できます。 Feature の色は設定可能で、塩基のナビゲートおよび選択するために使用できます。
その他
- “Range of Bases” を選択: 選択する塩基番号を入力して塩基を選択
- クラスタリングの速度と精度の向上
- Feature (タグ) カラーの変更
- プロジェクトビューでサンプルコメントの編集
- プロジェクトの特定の feature (タイプ) のみを表示できる新しい feature (タグ) 設定
- シーケンス検索時のあいまいさの処理の改善
CodonCode Aligner 9.0 は、Windows 10 と macOS 10.14 (Mojave) に対応しています。
