クイックツアー
このクイックツアーでは、CodonCode Alignerの基本的な機能を紹介します。具体的には、新規プロジェクトの作成、サンプルの追加、エンドクリップ、アセンブル、いくつかの編集機能について紹介します。チュートリアルで利用するサンプルデータをご希望の場合には、soft.support@hulinks.co.jp までご連絡ください。
※CodonCode Aligner 中で何かを選択したり、クリックするものについては「」を付けています。

1.CodonCode Aligner を起動します。
2.ここでは、新規プロジェクトを作成します。ラジオボタンで「Create a new project」を選択し、「OK」をクリックします。これで新規のプロジェクトが作成されました。


3.次に、プロジェクトにシークエンスを追加します。ABIまたはSCFフォーマットのクロマトグラムファイルや、様々なフォーマットのテキストファイルを追加することができます。ドラッグアンドドロップでもファイルやフォルダを追加できますが、ここではFileメニューからサンプルのフォルダを追加する方法を紹介します。
4.メニューバーから「File→open…」を選択します。ここでは、CodonCode Alignerがインストールされているフォルダに含まれているExample Filesフォルダで、「すべてのファイル」を選択し「Example1.ccap」というファイルを開きます。


5.Project View:Alignerのproject viewでは、エンドクリップとアセンブリをすることができます。さきほど開いたサンプルはUnassembled Samplesフォルダに追加されています。左の「▶」をクリックして展開します。


6.Qualityの列ではクオリティスコアが20以上の塩基が配列の中にいくつ含まれているか表示しています。

7.列のヘッダーをクリックすると行を並べ替えることができます。例えば、「Length」をクリックすると、Lengthの大きさで昇順に並び替えられます。もう一度クリックすると、降順に並び替えられます。


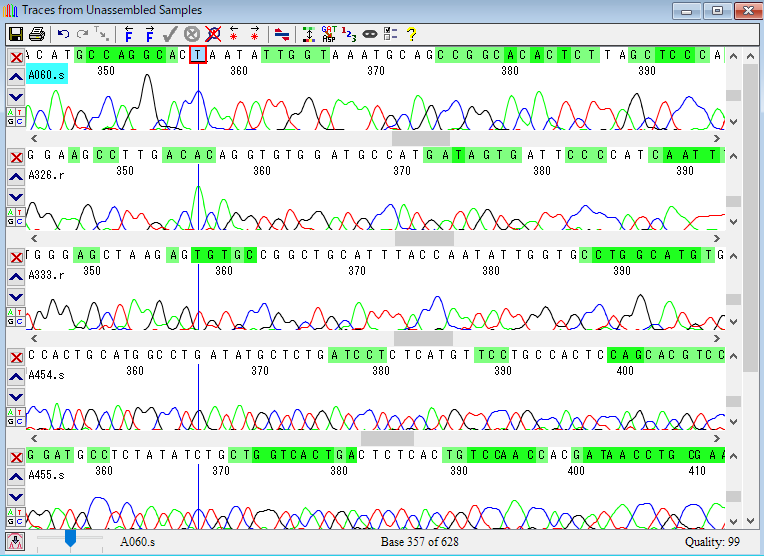
8.トレースを見る:いくつかの配列のクロマトグラムを見てみましょう。図10のウィンドウの「Unassembled Samples」をダブルクリックしてトレースを開きます。トレースの緑でハイライトされている部分は配列のクオリティが低いことを示しています。


9.次に、スクロールの方法、トレースの拡大・縮小の方法、塩基を変更する方法を紹介します。スクロールは、スクロールバーで行うことができます。デフォルトの配色では、3つの異なるクオリティを表しています(白:high quality、薄い緑:medium quality、濃い緑:low quality)。他にも違う配色を使うことができます。

10.図13のウィンドウの上部にある「配色切り替えアイコン」をクリックします。すると、図14のような配色に変わります。これは、連続的配色で、緑のハイライトが濃いほどクオリティが低いことを表しています。


11.もう一度「配色切り替えアイコン」をクリックします。図15の配色は、塩基を表しています。

12.もう一度「配色切り替えアイコン」をクリックします。図16の配色は、アミノ酸を表しています。再度「配色切り替えアイコン」をクリックすると、最初の配色に戻ります。

13.塩基を変える:塩基を選択し、新しい塩基を打ち込むことで塩基を書き換えることができます。これは、塩基が表示されているすべてのウィンドウで実行可能です。図17の「赤枠で囲ったC」を選択し、Tを打ち込んで塩基を変えてみます。

14.塩基を書き換えられることを確認したら、「Edit→元に戻す Base Edit」をクリックして変更した塩基をもとに戻します。
15.右の内側のスクロールバーでは、縦軸の倍率を変えることができます。また、左下のスライダーでは、横軸の広がりを変えることができます。

16.トレースをとがらせる:ピークが重なり始める領域で作業する場合、トレースをとがらせることができます。図20の左下の「Show sharpened tracesボタン」をクリックします。この方法でとがらせたトレースは、人為的な結果であることに留意してください。再度左下の「Show sharpened tracesボタン>」をクリックするともとのトレースに戻ります。


17.図22のウィンドウの右側にある外側のスクロールバーで、現在見えていないトレースを見ることができます。スクロールバーで隠れているトレースを見られることを確認したら、図22のウィンドウを閉じます。

18.エンドクリップ:配列をコンティグにアセンブルする前に、エンドクリップをすると良いでしょう。エンドクリップは、配列の初めと終わりから、クオリティが低い配列を取り除く作業です。図23でLengthとQualityの列を見てみると、それぞれの配列には約200~400個のクオリティの低い塩基があることが分かります。(Qualityの列ではクオリティスコアが20以上の塩基が配列の中にいくつ含まれているか表示しています。)

19.「Sample→Clip Ends…」をクリックします。エンドクリップの前に、図24のウィンドウでAlignerは何が切り取られるのかの概要を示します。ここでは、平均8個の塩基が配列の初めから、平均191個の塩基が配列の終わりから切り取られます。図24のウィンドウのChange Parametersでは、エンドクリップの厳密さを変えることができ、それが切り取られる塩基の数にどう影響するのか見ることができます。

20.Change parametersの設定は変えずに、図24の「Clip」をクリックします。すると、図25のようにLengthとQualityの値が近くなります。

21.図25の「Unassembled Samples」をダブルクリックしてトレースを開きます。すると、図26のように、配列の初めの部分が切り取られています。横スクロールして配列の終わりの部分も見てみましょう。図27のように配列の終わりも切り取られています。図27のウィンドウを閉じます。


22.プロジェクトを保存する:アセンブルをする前に、プロジェクトを保存しましょう。「File→Save Project As…」をクリックして、Example1という名前で保存します。
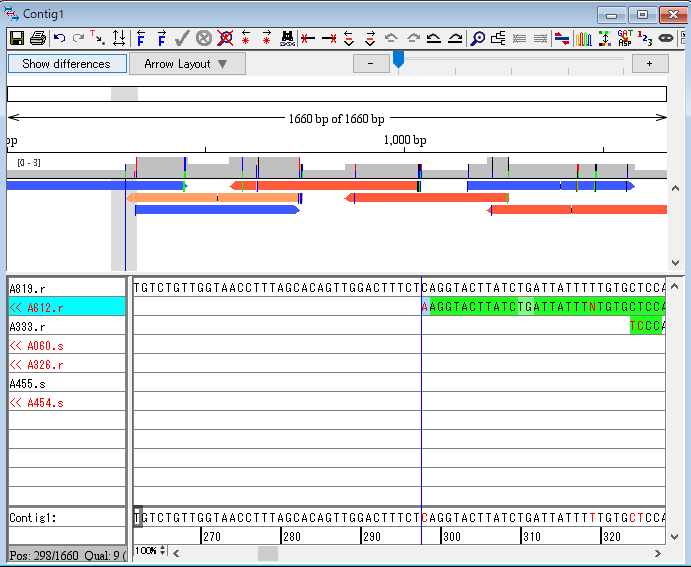
23.アセンブル:次に、サンプルをコンティグにアセンブルして、結果を見てみましょう。「Unassembled Samples」を選択した状態で「Contig→Assemble」をクリックします。図28の赤枠で囲ったContig1が新しく追加されたコンティグです。左の「▶」をクリックして展開します。


24.The Contig View:Contig Viewでは整列された塩基が表示され、コンティグの編集ができます。図29のウィンドウの「Contig1」をダブルクリックします。

25.図31の赤枠で囲った部分は配列の概観図です。概観図のどこかをクリックすると下に、配列のどの部分かが表示されます。今回は、図31の概観図中の「矢印で示した箇所」をクリックします。


26.図33の赤枠で囲った文字が赤色になっている部分に注目してください。デフォルトでは、CodonCode Alignerは最もクオリティの高い塩基をコンセンサス配列の塩基とします。この部分では、Tがそれに値します(緑のハイライトはクオリティが低いことを表しています)。「赤丸で囲ったT」をクリックしてトレースを確認してみましょう。


27.図34のウィンドウを閉じます。図35のウィンドウの右上にある「 マスクアイコン
」をクリックして、コンセンサス配列と違う塩基のみを表示させてみましょう。確認したら、もう一度「マスクアイコン」をクリックしてもとに戻します。


28.コンティグの編集:次に、コンティグを編集する方法を紹介します。すべての機能はSampleメニューまたはContigメニューから利用できますが、今回は、ツールバーボタンを使います。(macOSでは、ツールバーボタンがテキストと一緒に表示されよりスペースを取るため、今回使うアイコンが見えない可能性がありますが、ツールバーのPreferencesからテキストを表示するかしないかを、カスタマイズできます。)
29.図37の「赤丸で囲ったC」をクリックして選択状態にし、メニューバーから「Sample→Insert Gap→Shift Bases Right」をクリックします。選択した箇所にギャップが入ったことを確認したら図38の左上にある「Undoアイコン」でもとに戻します。


30.次に、図39のウィンドウの上部中央にある「Insert gap, shifting bases to the leftアイコン」をクリックして、先ほどとは逆側に配列がシフトしてギャップが入ることを確認したら、「Undoアイコン」でもとに戻します。

31.コンセンサス配列の編集をすると、すべてのサンプルが編集されます。塩基を挿入するには、まずギャップを挿入し、塩基を打ち込みます。まず、図40の「赤丸で囲ったT」をクリックして選択状態にし、「Insert gap, shifting bases to the rightアイコン」をクリックしてギャップを挿入します。


32.先ほど作ったギャップに「A」と打ち込みます。コンティグを編集できたことを確認したら、「Undoアイコン」を2回クリックして、もとに戻します。

33.塩基の削除:塩基を削除するには、deleteキーかbackspaceキーを押します。また、配列の初めからカーソルまで、または、カーソルから配列の終わりまでのすべての塩基を削除することもできます。この操作は、サンプルでもコンティグでも行うことができます。
34.図43の「赤丸で囲ったC」をクリックし選択状態にして、「Sample→Delete→From Sample Start」をクリックします。選択したCから左側の配列が削除されていることを確認したら「Undoアイコン」でもとに戻します。

35.次に、図45の中央上部にある「Delete all bases to the rightアイコン」をクリックします。選択したCから右側の配列が削除されていることを確認したら「Undoアイコン」でもとに戻します。


36.図47の「赤丸で囲ったT」をクリックし選択状態にして、「Contig→Delete→From Contig Start」をクリックします。コンティグが削除されたことを確認したら、「Undoアイコン」でもとに戻します。


37.コンティグの編集:次に、コンティグを編集する方法を紹介します。すべての機能はSampleメニューまたはContigメニューから利用できますが、今回は、ツールバーボタンを使います。(macOSでは、ツールバーボタンがテキストと一緒に表示されよりスペースを取るため、今回使うアイコンが見えない可能性がありますが、ツールバーのPreferencesからテキストを表示するかしないかを、カスタマイズできます。)


38.サンプルの削除と追加:次に、読み込んだ配列をコンティグから取り除き、コンティグへ追加する方法を紹介します。図51の「矢印で示した箇所」をクリックして、「赤丸で囲ったG」を右クリックします。出てきたメニューから「Move to Unassembled Samples」をクリックします。図52のウィンドウを消すと、図53のように、先ほど取り除いたサンプルがUnassembled Samplesに移動しているのが分かります。



39.除外されたサンプル (A612.r) をContig1に戻す (追加する) には、Contig1にサンプル (A612.r) をドラッグ&ドロップします。

40.PreferencesとHelp:このクイックツアーを終える前に、設定の方法とオンラインヘルプの使い方の2つを紹介します。
41.まず、「Edit→Preferences…」をクリックします。CodonCode Alignerでは様々な設定を変えることができます。例えば、図55のウィンドウでは塩基の色を変えることができます。どの設定を変えるかは、左側のリストをクリックすることで選択できます。

42.もう一つ設定の例を示します。図56のウィンドウの左側のリストから「Assembly」をクリックします。図56のウィンドウでAlgorithmを変更します。「End to end alignments」のドロップダウンメニューを開いて、「Large gap alignments」をクリックすると、cDNAをゲノムDNAに並べ替えることができます。


43.次に、図58のウィンドウの左下にある「Help」をクリックします。左側にあるパネルを使って様々なヘルプを見ることができます。図59のウィンドウの左上の3つのアイコンをクリックすると、左からコンテンツ、インデックス、検索モードに切り替えることができます。


44.図59のウィンドウの左上の3つのアイコンのうち、中央にある「インデックスアイコン」をクリックします。

45.次に、図59のウィンドウの左上の3つのアイコンのうち、右側にある「検索モードアイコン」をクリックします。

46.図61のウィンドウとPreferenceウィンドウを閉じます。図62の赤枠で示したHelpメニューやF1キーからでもヘルプにアクセスできます。

47.以上でCodonCode Alignerのクイックツアーを終了します。
その他のチュートリアルは、こちらのウェブサイトをご覧ください。
