CodonCode Aligner 入門
このチュートリアルでは、CodonCode Aligner のいくつかの機能を紹介します。具体的には、新規プロジェクトの作成、サンプル追加、整列、編集、コンティグの比較、いくつかの解析機能を紹介します。チュートリアルで利用するサンプルデータをご希望の場合には、soft.support@hulinks.co.jp までご連絡ください。
※CodonCode Aligner 中で何かを選択したり、クリックするものについては「」を付けています。

1.CodonCode Aligner を起動します。
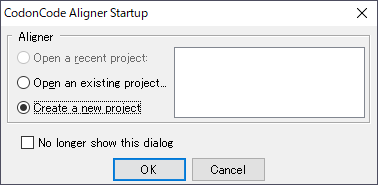
2.ここでは、新規プロジェクトを作成します。「Create a new project」を選択し、「OK」をクリックします。
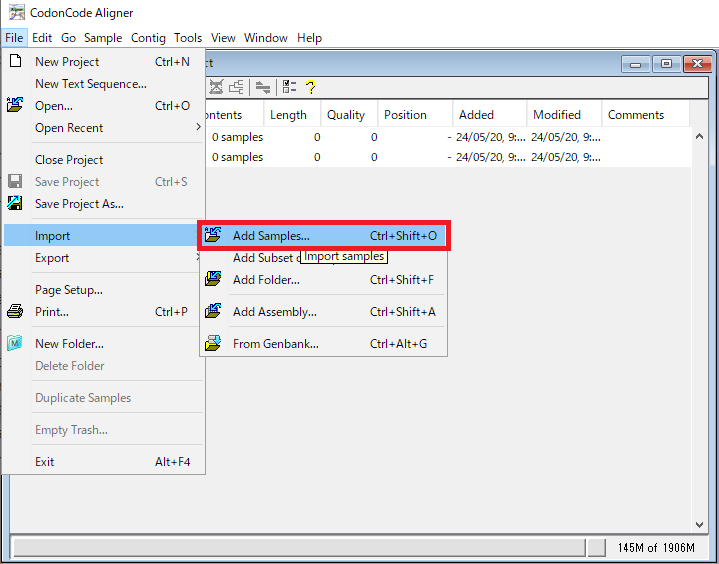
3.プロジェクトにシークエンスを追加します。ABI や SCF フォーマットのクロマトグラムファイルや各種フォーマットのテキストファイルの追加が可能です。「File → Import → Add Samples…」を選択します。
4.今回は、リファレンス配列を使います。リファレンス配列がなくてもサンプルを構築することができます。ここでは「IntroductionToAligner」フォルダを開き、ファイルのタイプですべてのファイルを選択し、「Reference.fasta」ファイルを開きます。

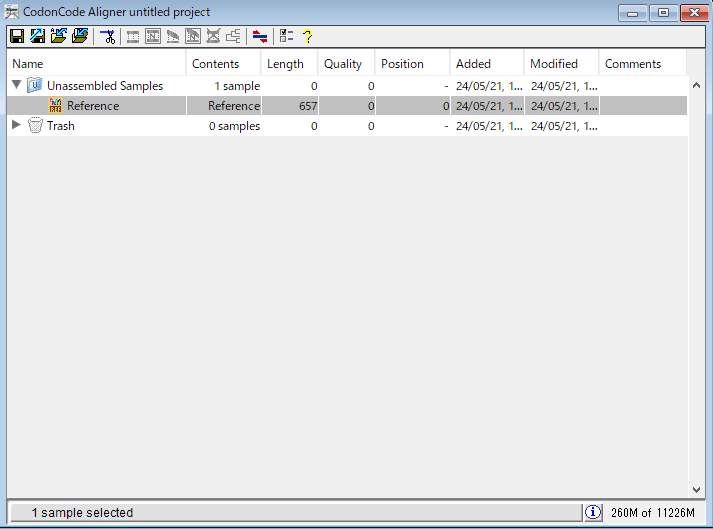
5.Reference というファイルがリファレンス配列です。次に、Aligner にそれを認識させ、適切にマークします。

6.「Reference」を選択して「Sample → Make Reference Sequence」をクリックします。すると、配列の名前の左側のアイコンと Contents の表示が変わります。これで、Aligner がこの配列をリファレンス配列だと認識しました。
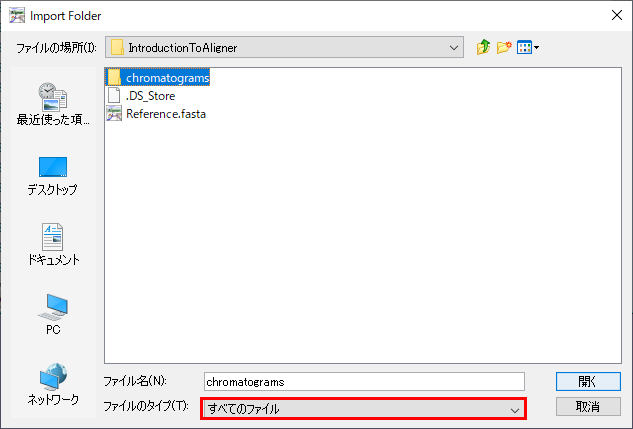
7.次に、クロマトグラムのフォルダを追加します。「File → Import → Add Folder…」を選択します。IntroductionToAligner フォルダから「chromatograms」というフォルダを選択して開きます。
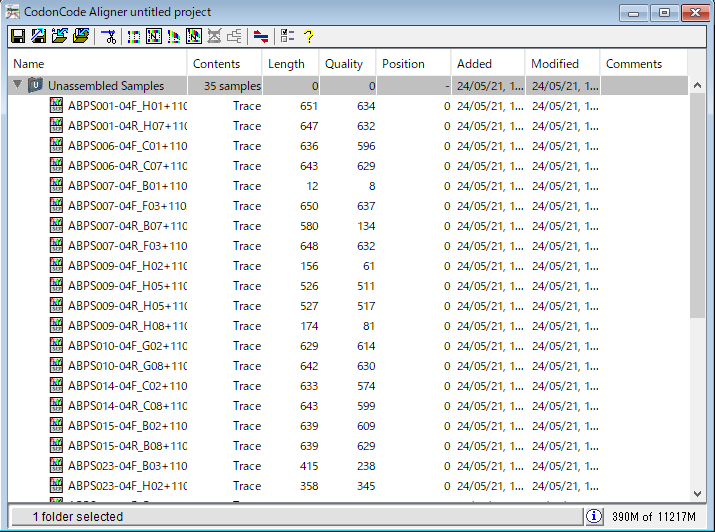
8.インポートしたクロマトグラムは 12種類の小型哺乳類のものです (ナキウサギ属)。次に、それぞれの種のフォワード配列とリバース配列をサンプル名により自動的に整列させます。「Contig → Advanced Reference Alignments → Align in Groups…」を選択します。図8 のダイアログで「Cancel」を選択します。
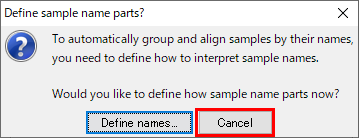
9.サンプル名がどのように解釈されるのか定義する必要があります。図9 のウィンドウで「Define name parts…」をクリックします。
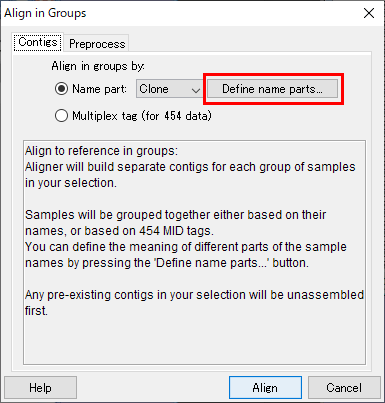
10.図10 のウィンドウで「Guess name scheme」ボタンをクリックして、Aligner に名前の配置を推測させます。
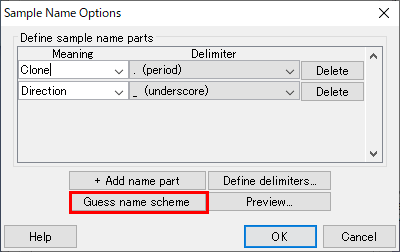
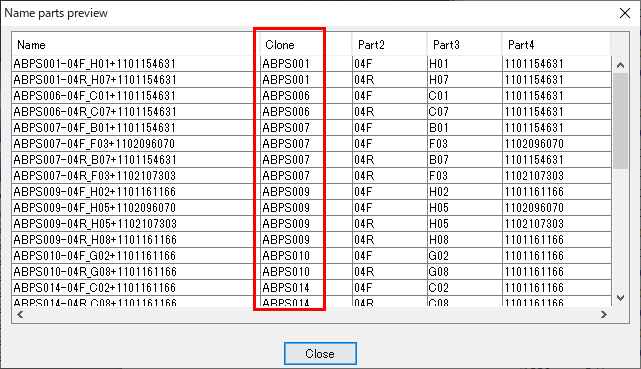
11.名前の配置を確かめるために、サンプル名がどのような語法になるのかを表す「Preview…」をクリックして確認します。図13 のウィンドウでは、Aligner が名前を分けて、初めのパートを “Clone” として分類しています。
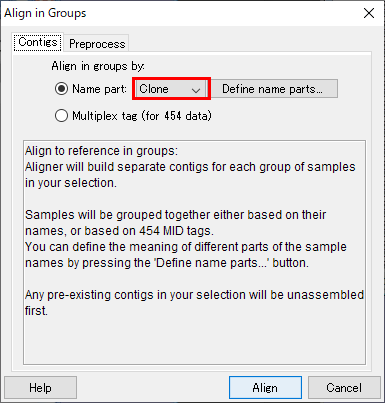
12.名前の配置を自分で定義することもできます。図13のウィンドウを閉じて、図12のウィンドウも「OK」をクリックして閉じます。そして、図14のウィンドウの Name part: の右にあるドロップダウンメニューが「Clone」になっていることを確認して、「Align」をクリックします。すると、それぞれの種に対し一つの 12個のコンティグが作られます。
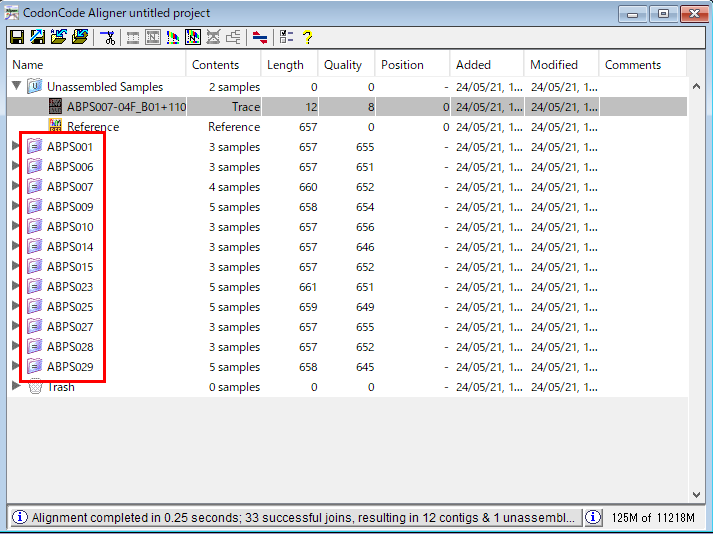
13.コンティグの一つを見てみましょう。「ABPS001」をダブルクリックすると、contig view が開きます。contig view にはズームバー、範囲地図、整列させた配列の概観図、整列させた塩基配列とコンティグ(コンセンサス)配列が表示されます。
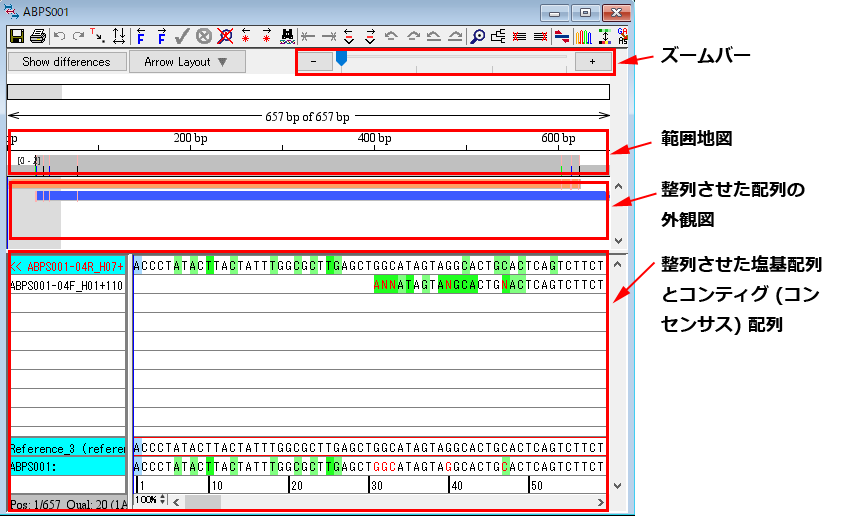
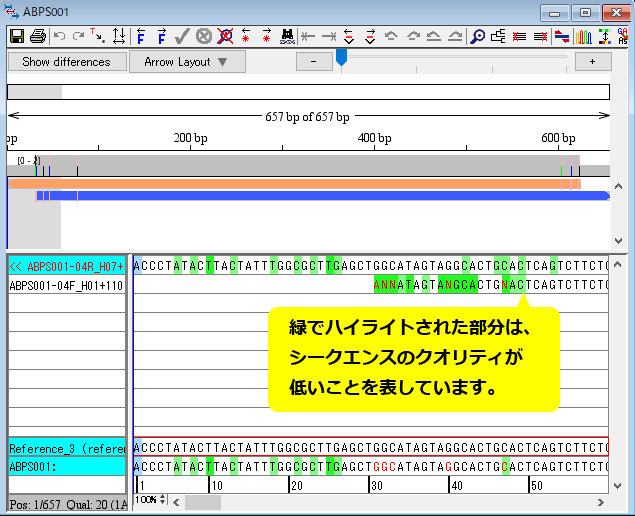
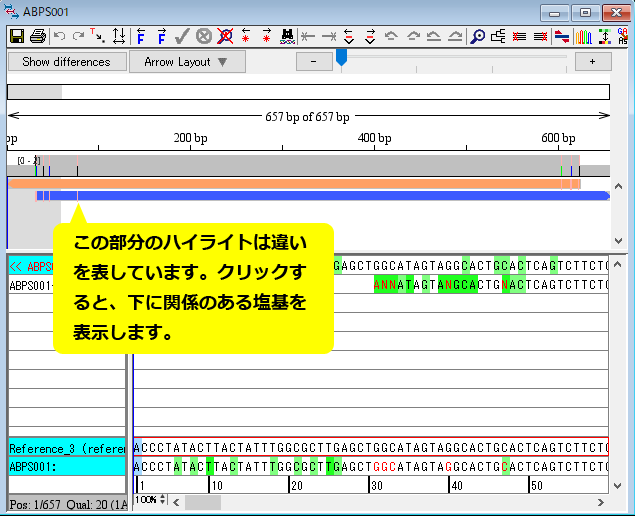
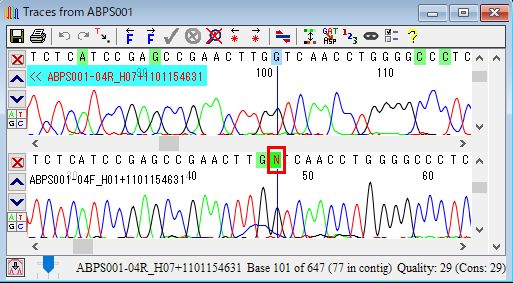
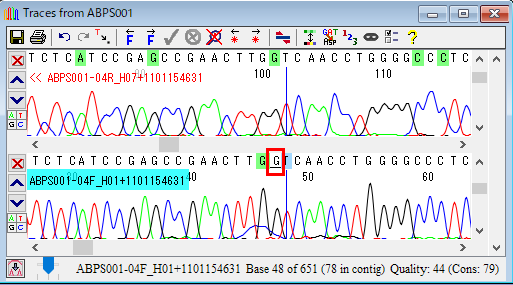
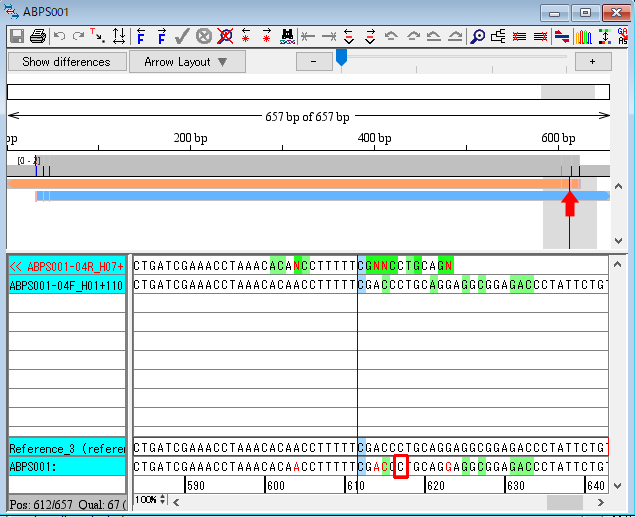
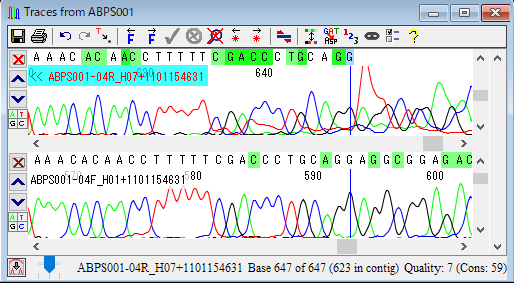
14.整列させた配列の概観図の「青い帯のハイライト」をクリックして、違いを見てみましょう。「赤丸で示した塩基」をダブルクリックして trace view を開きます。trace view の塩基は手動または自動で編集可能です。手動の場合は、塩基を選択して打ち込むだけで編集できます。
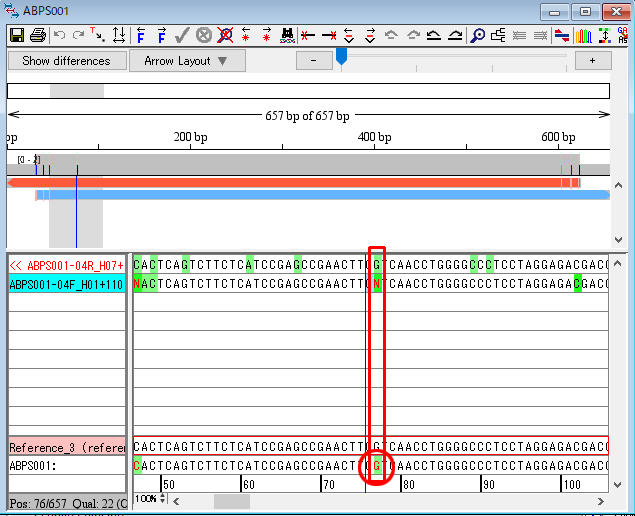
15.trace window を閉じます。今度は、contig view の「オレンジの帯の右側、3本のハイライトのうち真ん中の線」をクリックします。そして、「赤枠で囲った C」をダブルクリックします。
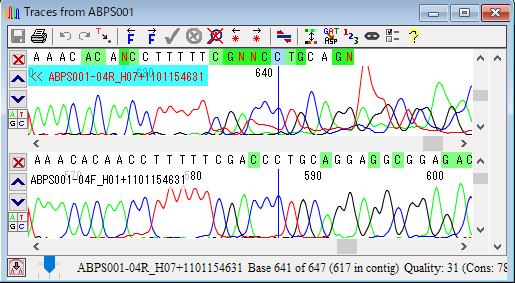
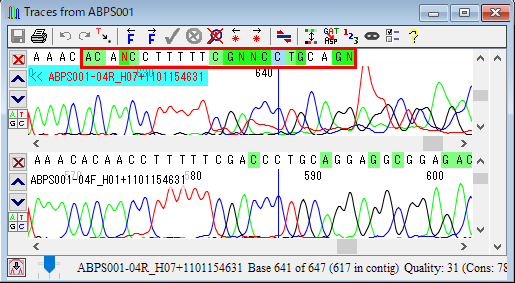
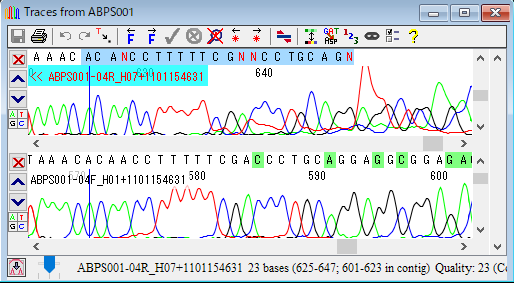
16.塩基を自動的に編集することもできます。例えば、コンセンサス配列と一致させるように編集できます。赤枠で囲ったところをドラッグして選択状態にして、「Edit → Change Bases → Match Consensus」を選択します。図27 のダイアログで「OK」をクリックします。すると、コンセンサス配列と一致するように、塩基が編集されました。
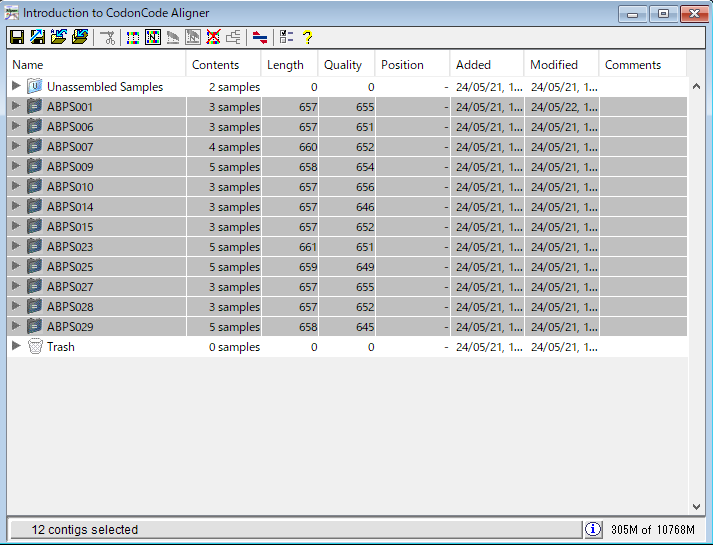
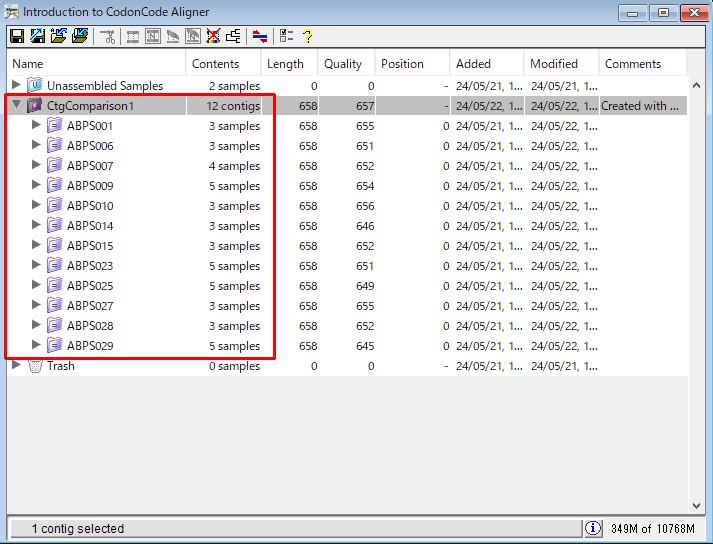
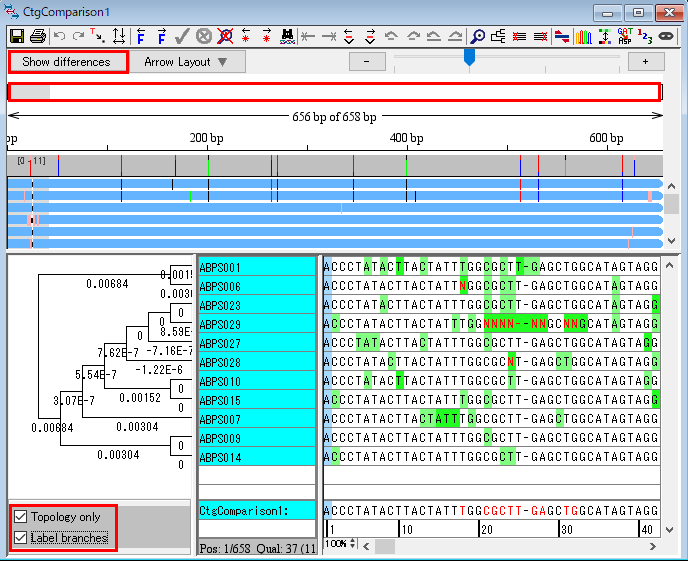
17.このチュートリアルでのゴールは種間での配列の違いを見つけることです。CodonCode Aligner では、コンセンサス配列を複製またはエクスポートすることなくコンティグを直接比べることができます。trace window と contig view を閉じます。ABPS001 を選択した状態で ABPS029 を Shift+ クリックして、「ABPS001 から ABPS029 まですべて」選択します。そして、「Contig → Advanced Assembly → Compare Contigs…」をクリックして図30 のウィンドウを開きます。
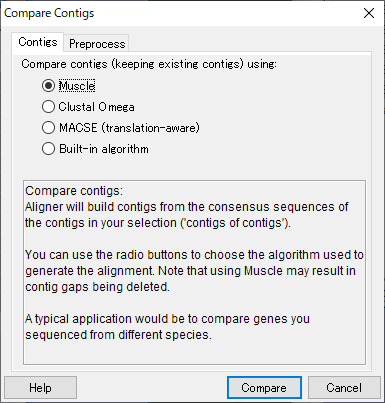
18.図30 のウィンドウでラジオボタンが Muscle に選択されていることを確認し、「Compare」をクリックして、図31 のウィンドウで「OK」をクリックします。これで、コンティグの比較が作られました。違う種同士のためのコンティグの整列です。
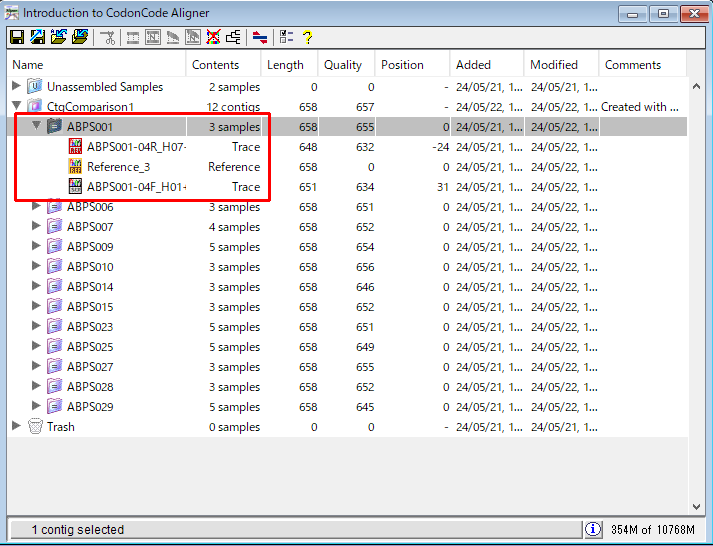
19.ABPS001 の左の「▶」をクリックして展開します。それぞれの独立したコンティグは下部のトレースへのリンクを保持しています。これにより違いを確かめたり編集したりするのがとても簡単になります。
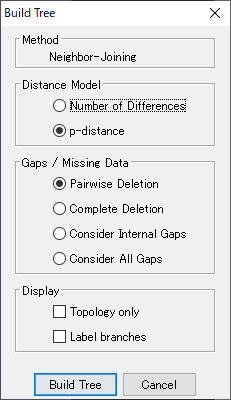
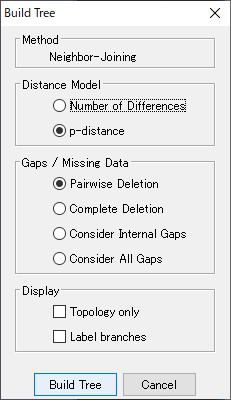
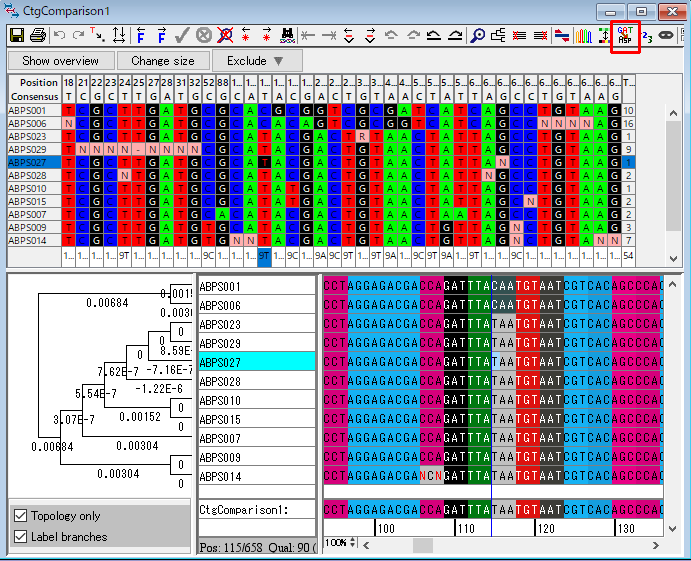
20.ABPS001 と CtgComparison1 の展開を閉じて、「CtgComparison1」をダブルクリックします。種ごとにどのような違いがあるか調べるために系統樹を作成します。右上の「系統樹アイコン」をクリックして、図35 のウィンドウで設定は変えずに「Build Tree」をクリックします。すると、系統樹が作成されます。
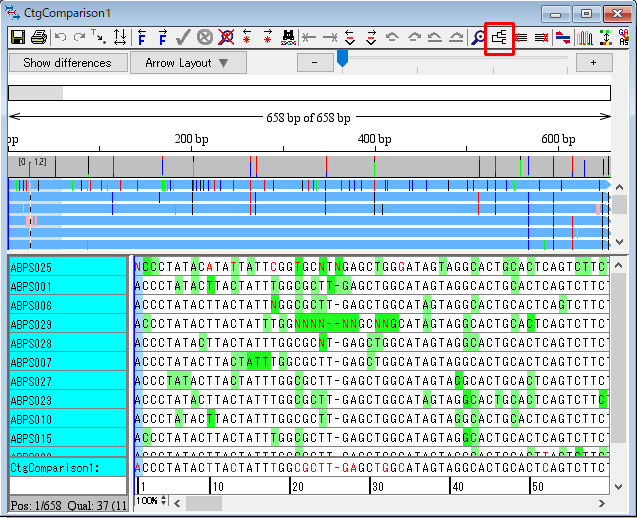
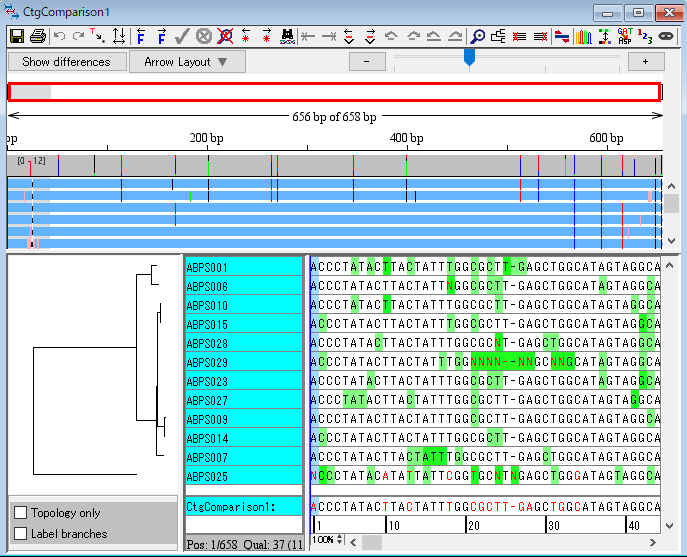
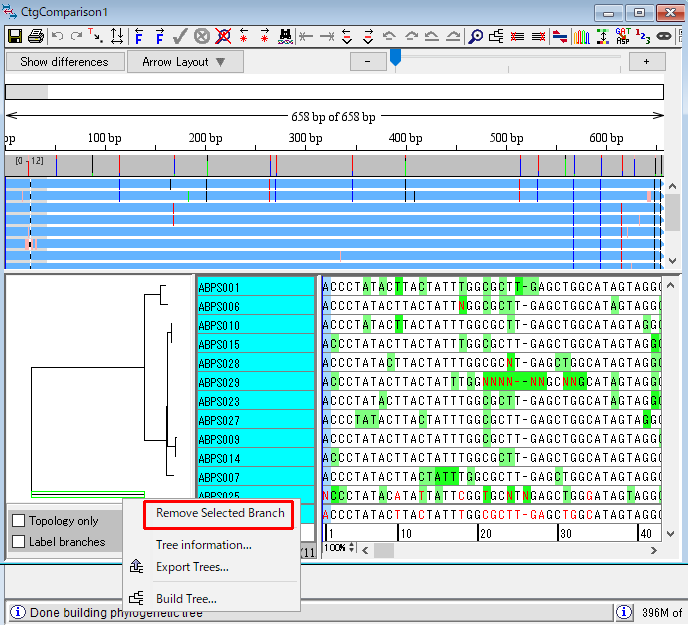
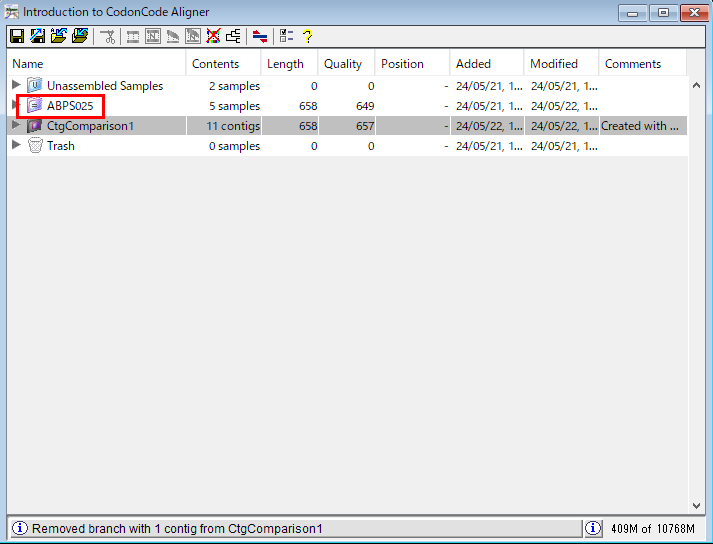
21.ABPS025 という配列だけほかの配列とずいぶん違います。サンプルを取り違えたのでしょうか?この配列をこのコンティグ比較から取り除きましょう。「赤枠で囲った系統樹の枝」を右クリックして、「Remove Selected Blanch」を選択します。図39 のダイアログで「OK」をクリックしたら、CtgComparison1 ウィンドウを閉じます。すると、ABPS025 が CtgComparison1 の外に取り除かれています。

22.配列の品質コントロールとして系統樹の枝を取り除く機能を使うことができます。「CtgComparison1」をダブルクリックします。系統樹の枝を取り除くのと同様に系統樹を削除して、新しい系統樹を作りましょう。contig view で右上の「系統樹アイコン」をクリックし、図43 のウィンドウで設定を変えずに Build Tree ボタンを押して系統樹を作成します。
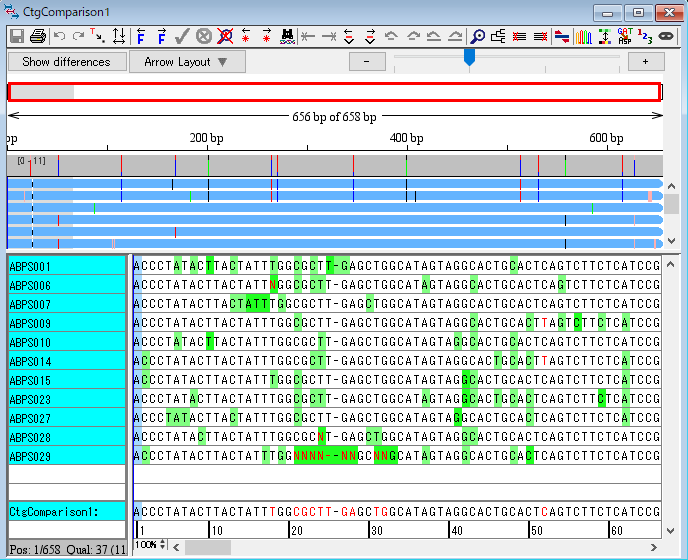
23.作成された系統樹の下にある「Topology only」と「Label branches」のチェックボックスをオンにします。contig view は概観かまたはそのコンティグの difference table を表示することができます。左上の「Show difference」ボタンをクリックします。
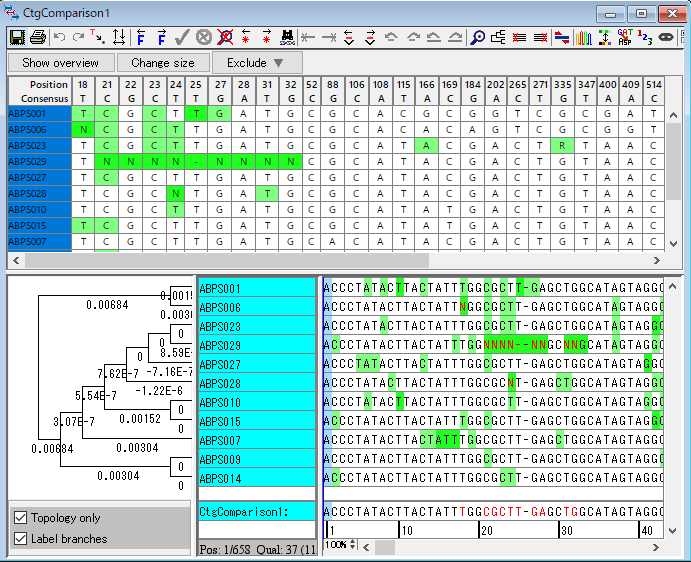
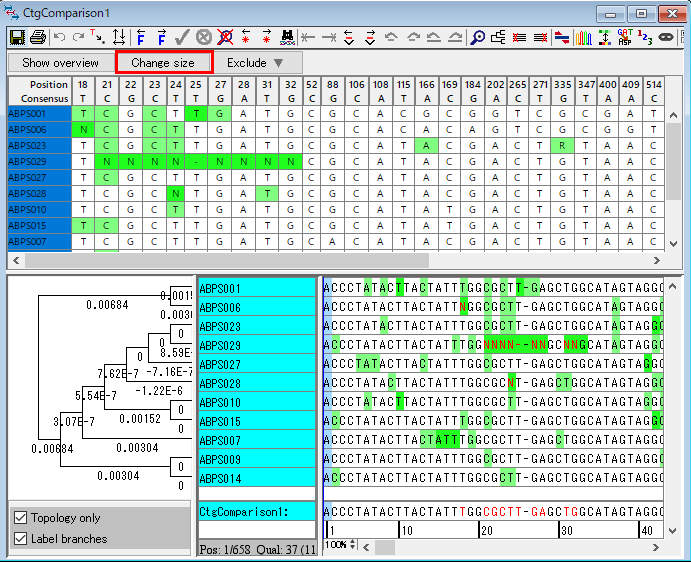
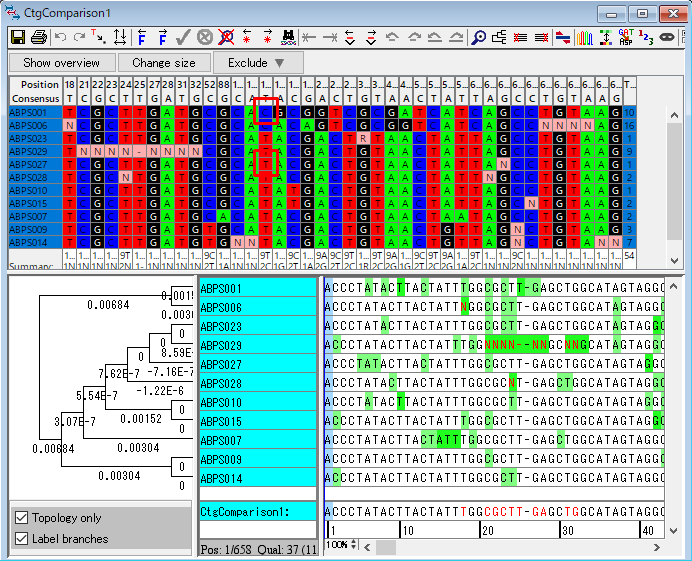
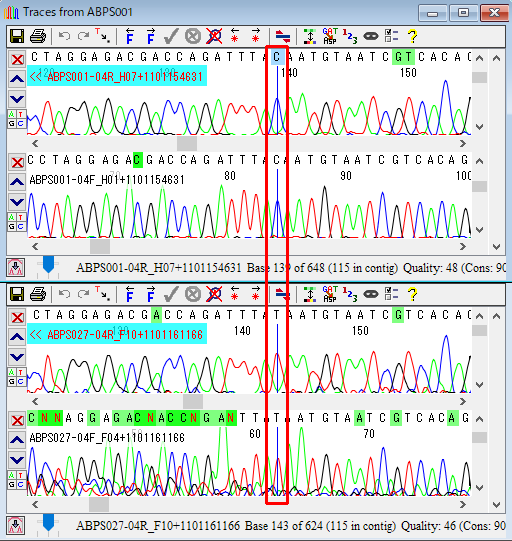
24.左上の「Change size」ボタンをクリックします。difference table のどの塩基をクリックしても、コンティグの中で一致する塩基を見ることができます。今回は、図47 のウィンドウの「赤枠で囲った C と T」をダブルクリックして、trace window を開きます。開いた2つのウィンドウを比較して下さい。
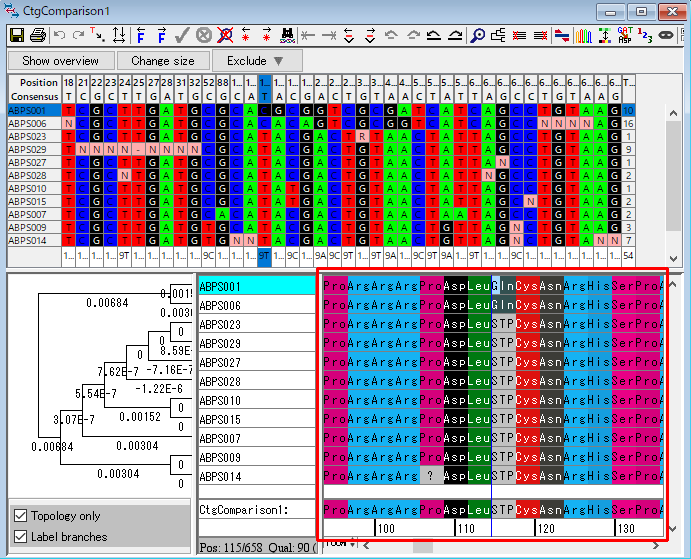
25.2つの trace window を閉じます。アミノ酸と翻訳に基づくバックグラウンドを見ることにより、アミノ酸の変化を見つけることができます。「View → Sequence Colors → Translation Based」を選択します。右上の「GAT → ASP アイコン」をクリックすると、塩基配列がアミノ酸配列の表示に変化します。
26.本チュートリアルはこれで以上です。CodonCode Aligner にはほかにも以下のような機能があります。
- 制限マップと RFLP 解析
- プライマーの設計
- 変異探索と解析
- 複数の出力オプション
- ベクターの切り取りとエンドクリップ など
その他のチュートリアルは、こちらのウェブサイトをご覧ください。
