 |
| サイトマップ | |
|
 |
| サイトマップ | |
|
シーケンスの装飾 (Sequence Adornments) では、コピーしたいシーケンスエディタの範囲を Option キーとマウスドラッグを使用して、その一部を素早く簡単にキャプチャして、GI ノートブックや他のプログラムに画像として表示させる方法を説明しました。シーケンスデータを実際に使用して出力表示の代わりにしたい場合は、GI ノートブックに Features Object を作成します。Features オブジェクトには、一つまたは複数の核酸 (又はタンパク質) シーケンスを翻訳や切断部位インジケーターと共に含めることができます。その外観は、GI ノートブックの他のオブジェクトと同様に変更することができます。
| ※ b. シーケンス全体を選択して (シーケンス名をクリックして)、シーケンスウィンドウから直接ノートブックにドラッグすることも可能です。これにより GI ノートブックに Features オブジェクトが自動的に作成されます。シーケンスの一部しか選択せずに GI ノートブックにドラッグすると、それはテキストとして処理され、この場合、配置されるのはバックグラウンドテキストの一部となります。 |
まずはじめに、Features オブジェクトとして GI ノートブックに表示させたいシーケンスのセグメントをシーケンスエディタウィンドウから選択します。Edit > Copy を選択すると、シーケンスウィンドウからシーケンスがクリップボードbにコピーされます。コピーしたシーケンスを GI ノートブックにペーストすると、そのシーケンスを含む Features オブジェクトが自動的に作成されます。クリップボードに複数のシーケンスが存在する場合は、複数シーケンスの Features オブジェクトが作成されることになります。これについては後で説明します。
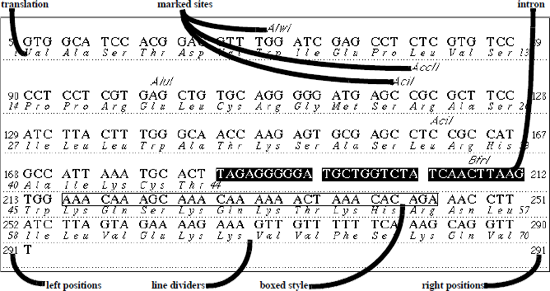
Features オブジェクトは、他の GI ノートブックオブジェクトと同様に GI ノートブック内でマウスを使って自由に動かすことができます。Features オブジェクトをダブルクリックして選択状態にすると、そのオブジェクトにシーケンスが一つしかない場合は、Features メニューが追加されます。Features オブジェクトに複数のシーケンスがある場合は、Sequences メニューが表示されます。図 3.14 は、DNA の Features オブジェクトの各部分をあらわしたものです。
 |
Features オブジェクトに編集を加えることはできませんが、その表示法は変更することができます。GI ノートブックに配置した後は、シーケンスに変更を加えることはできません (シーケンスをあらわす文字列を含むオブジェクトではありますが、本当のシーケンスではありません)。Features オブジェクトは、GI ノートブックの一部となった時点でオリジナルのシーケンスとの関連が失われることになります。シーケンスの中の強調表示したい特徴 (feature) をハイライトで表示するために Features オブジェクトが使用されます。
Features オブジェクト内のシーケンスに様々な操作を実行する場合は、Features メニューを使用します。Mark Sites... メニューオプションを使えば、DNA の制限酵素部位や、タンパク質の化学/制限酵素切断部位にマークを付けることができます。この選択肢は、Restriction enzyme digest (制限酵素消化) や Protein cleavage (タンパク質開裂) 解析の実行を選択した場合と同じです。Translate... , Define Intron, Undefine Intron(s) は DNA の Features オブジェクトのみで利用できる選択肢です。Translate... を選択すると、DNA 配列の下に指定した翻訳テーブルを使用したアミノ酸配列が作成されます。DNA のあるセグメントがイントロンとして指定されている場合は、そのセグメントは DNA の翻訳でスキップされますc。
| ※ c. Features オブジェクトに翻訳によってアミノ酸配列が作成されると、それはプレーンテキストとしてシーケンスエディタにコピー&ペーストし、各種操作を実行することができます。なお、シーケンスウィンドウにシーケンスをコピーする前に、Features オブジェクトでは翻訳を1文字アミノ酸 (one letter amino acids) で表示させます。 |
DNA のあるセグメントをイントロンに指定するには、そのセグメントを選択状態にしたあと、Features > Define Intron を選択します。DNA シーケンス内では任意のイントロンを指定することができます。同じ DNA から、例えば、読み枠 (reading frame) の違いによって、複数の翻訳をすることもできます。
Features > Display サブメニューを使えば、Features オブジェクトの見た目を思い通りに指定することができます。Site Markers (制限酵素部位やタンパク質切断部位のサイトマーカー) 、Left Positions (左端の番号)、Rignt Positions (右側の番号)、Line Dividers (シーケンス行間に表示する区切り線) の表示または非表示を選択することができます。また、翻訳された DNA の Features オブジェクトの場合は、翻訳 (Translations) の表示と非表示を切り替えたり、翻訳を One Letter AA Code (1文字アミノ酸コード) または Three Letter AA Code (3文字アミノ酸コード) のいずれかで表示することができます。Line Spacing を使用すると、各シーケンスの行間隔を指定することができます。
ポジション (位置) インジケーターのフォント特性を変更するには、その位置番号を選択してから Format メニューを使用して変更します。DNA シーケンスで翻訳されたシーケンスのポジションインジケーター (アミノ酸位置の番号) では、最も近くにあるアミノ酸文字の書式と一致するようにアミノ酸の番号の書式が設定されます。Format メニューからアミノ酸の番号フォント属性を変更することはできません。
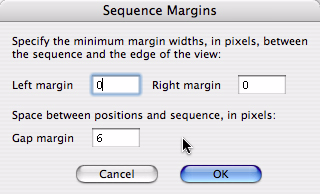
Features > Display > Features Margins... を選択すると、図 3.15 に示すダイアログが表示されます。このダイアログでは、シーケンス文字の端と、Features オブジェクト自身の境界線との間の間隔の最小値を設定できます。また、シーケンスとポジションインジケーターの間隔も設定することができます。
 |
Features > Grouping は、シーケンスのリスト表示で文字編成を設定するのに使用します。このサブメニューを使えば、シーケンスのセグメントを編成するグループのサイズと、改行の挿入や削除を定義することができます。
上記に説明した機能に加えて、Format メニューの中にあるオプションを使って、その書式を細かく調整することができます。この中で良く使うのは、シーケンス内で選択した任意のセグメントの周囲にシンプルなボックスを配置する Format > Style > Box Around 項目です。これは、Features オブジェクト内の特定のシーケンスに注意を向けさせたい場合に使用すると便利です。Box Around は、Style サブメニューの中にある項目と同じ働きをします。
GI ノートブックに多重シーケンスの Features オブジェクトを作成すると、Sequence メニューが表示されます。多重シーケンス整列にカスタム装飾 (custom adornments) を適用することはできますが、単一のシーケンスしかない場合に表示される Mark Sites... は、ここでは実行することができませんカスタム装飾については、こちらをご覧ください。
シーケンスエイディタ・ウィンドウは、シーケンスを操作する目的で設計されていますが、これとは反対に Features オブジェクトはシーケンスを表示する目的で設計されています。