| ※ 2つのユーザー事例「StarDropの予測モデルによる抗炎症剤の改良 」、「高速SAR解析でカナダの化学者を支援」を掲載しました |
創薬を成功に導くための完全なプラットフォーム
StarDrop™ は、最適なバランスのとれた化合物をすばやく提供します。予測モデルによる最新データから次の合成ターゲットや研究対象に関する意思決定までのシームレスなフローを可能にし、探索プロセスのスピード、効率、生産性の向上を実現します。
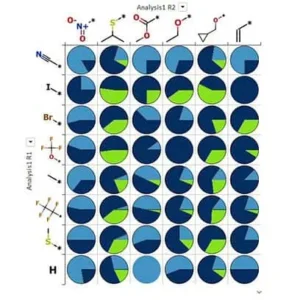
SAR 解析
R-グループ解析や、クラスタリング、Matched Pair Analysis (マッチドペア解析)、Activity Landscape Analysis (活性ランドスケープ解析) などの計算手法を用いて構造活性相関 (SAR) を解析することで、化合物の最適化を導く貴重な情報が得られます。StarDropは、これらの結果を視覚的に直感的な方法で表示し、誰もが結果を理解し、すばやく結論を導くことができるようにします。
データの可視化
StarDrop は、さまざまな可視化様式を瞬時に適用することができます。複数のグラフを同時に表示でき、そのすべてが相互にリンクされ、基になるデータにもインタラクティブにリンクされているため、複数のプロパティと複合構造間の関係を調べることができます。
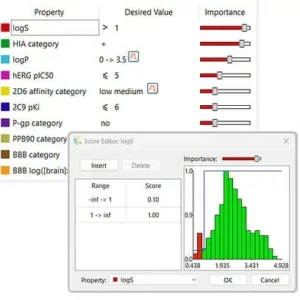
マルチパラメータ最適化
StarDrop は客観的で自信をもった決定を手助けします。これはマルチパラメータ最適化に向けたユニークなアプローチで、あなたのプロジェクトに合わせて化合物の優先順位をつけるために、多数のソースから得たデータを統合します。その際、価値のある機会を逃さないように、各データに固有の誤差も考慮します。 高い質を持つ薬物は、ターゲットに対する活性や ADME、安全性等、バランス良く多数の物性値をもつべきですが、しばしば競争的な物性値を含むことや実験値や予測値のもつ誤差により、その予測は非常に挑戦的な課題となっています。StarDrop のユニークな確率的なスコアリングは、マルチパラメータ最適化を実行し、バランスが良い物性値をもつ化合物を特定します。
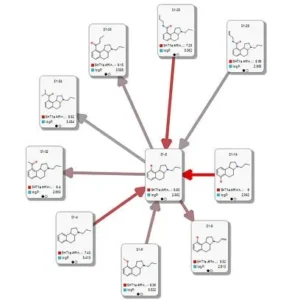
直感的な Card View®
Card View は、ユニークな方法で化合物を表示します。視覚的に、化合物間の関係を示し、最もバランスのよい化学シリーズや最適化の戦略を強調します。
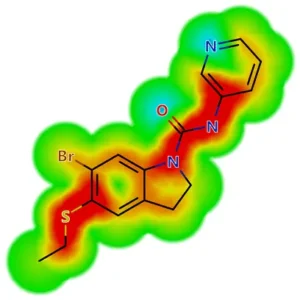
Dynamic Glowing Molecule™
Glowing Molecule (光る分子) の可視化は、予測された物性値に最も大きな影響を与える化合物の領域を強調表示します。これにより、より改良された物性値を持った化合物の設計を導きます。
シームレスな AI インサイト
Cerella AI プラットフォームの独自の機能を StarDrop 内から直接活用して、革新的なインサイトを明らかにします。
インフォマティクスインフラストラクチャとの統合
StarDrop の包括的なデータ解析、可視化、化合物設計環境は、CDD Vault、D360、Python スクリプティング、社内およびサードパーティの予測モデルなど、他のインフォマティクスインフラストラクチャとシームレスに連携するように構築されています。
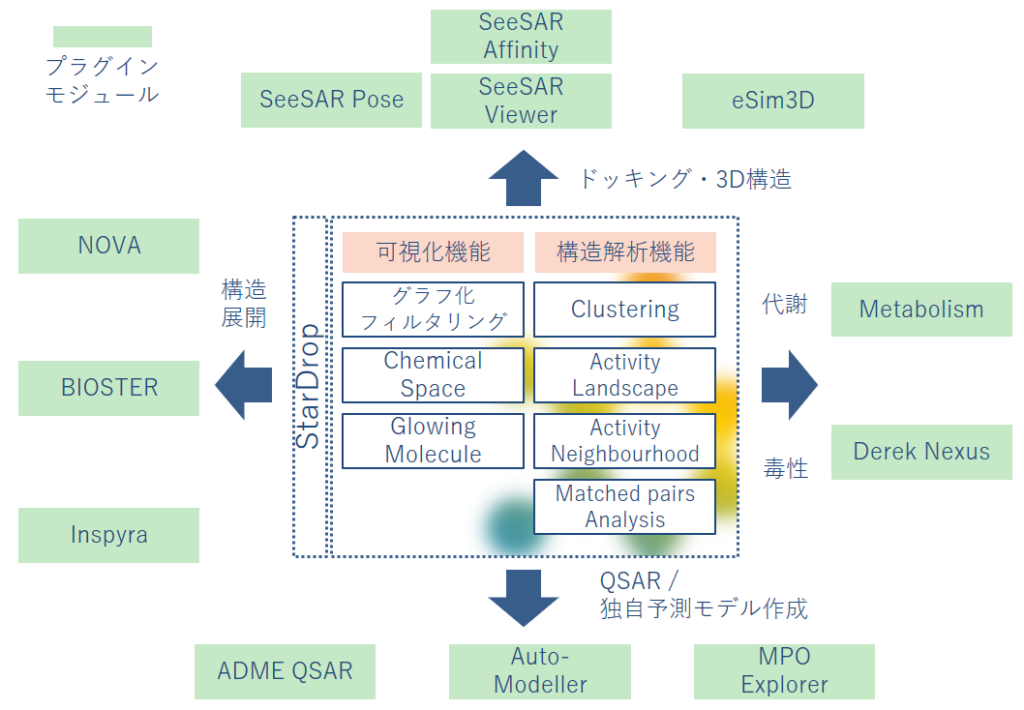
オプション
プラグインモジュール
インシリコモデリング、生成化学、3D 設計用のプラグインモジュールや、医薬品設計の拡張コラボレーション、社内外のデータベース統合など、幅広いカスタマイズや統合オプションで、必要なワークフローを構築できます。各モジュールの詳細は、こちらをご参照ください。
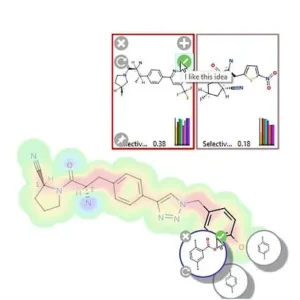
Idea Tracker
発見プロセス全体を通してすべての設計と決定をキャプチャします。StarDrop に記録されたすべての新規化合物の中央登録システムにより、プロジェクトデータを明確に可視化し、すべての関連情報を 1 か所にまとめることでチームの時間を節約します。一元化された共有で共同設計を促進し、機会の損失を回避し、創薬の効率を最大化します。
Pose Generation Interface
3D ドッキングおよびアライメントモデルと、創薬データの視覚化、2D SAR 分析、予測モデルを単一のエレガントな環境でシームレスにリンクします。これにより、設計の複数の反復を 3D でリアルタイムに評価できます。返されたポーズを調べることで、主要な結合の相互作用を理解すると同時に、実験やその他の予測方法からのデータも考慮することができます。
Query Interface
構造化されたデータベースクエリを作成、共有、実行するためのユーザーフレンドリーなツールです。視覚化と分析の準備が整った結果を返します。
開発元:Optibrium Ltd.
機能一覧
アップデート情報
各アップデート情報は こちら をご確認ください
動作環境
最小ハードウェア要件
Windows または macOS の場合:
- 4 GB RAM
- 2 GB ハードディスク空き容量
StarDrop サーバー (Model、Auto-Modeller™、License、Derek Nexus™ Web サービス) を Linux マシンにインストールする場合:
- 4 GB RAM
- 4 GB ハードディスク空き容量
Auto-Modeller サーバーのメモリ要件は、数千単位のデータポイントを含むデータセットをモデリングする場合、大幅に高くなる可能性があります。
StarDrop サーバーは、基盤プラットフォームの違いによる互換性問題を防ぐため、Docker コンテナに組み込まれたマイクロサービスとしてインストールされます。
Docker ベースのコンテナ化された StarDrop サーバーをインストールするには、Python 3.9 以降、Docker 20.10.24、Docker Compose 2.3.4 が必要です。
Metabolism サーバーの場合:
Metabolism サーバーは他のサーバーよりも計算負荷が高いため、別途インストールすることを推奨します。Metabolism サーバーをインストールするには、最新の CPU に加え、100 GB 以上の空きディスク容量 (/var パ ーティションを含む) と、以下のいずれかの構成が必要です。
- 最小:8 CPU cores、16 GB RAM
- 推奨:16 CPU cores、32 GB RAM
- 最適:32 CPU cores、64 GB RAM
OS
StarDrop 8.0 クライアント:
- Windows 11
- macOS 26 (Tahoe)
StarDrop サーバー (Model、Auto-Modeller、License サーバー)、Derek Nexus ウェブサービス、Metabolism サーバー:
64 ビット Linux オペレーティングシステム
- Ubuntu 24.04 LTS
- Red Hat Enterprise Linux 9 / 10
ライセンス
StarDrop デスクトップクライアント
StarDrop およびオプションの ADME QSAR、Nova、BIOSTER、Inspyra、SeeSAR、Surflex eSim3D、MPO Explorer モジュールはすべて Windows と macOS のデスクトップまたはノートパソコンで動作します。ご自分のパソコンに適したインストーラーをダウンロードして実行するだけです。
オプションの Metabolism、Auto-Modeller、Derek Nexus モジュールの計算を実行するにはサーバーが必要ですのでご注意ください。これらのモジュールは StarDrop インターフェースを通じてアクセスでき、必要なサーバーはセルフホストまたは Optibrium がホストすることができます。
StarDrop サーバー
StarDrop サーバーは、オプションの Metabolism および Auto-Modeller モジュールをサポートするか、フローティングライセンスを選択している場合のみ必要です。Derek Nexus のウェブサービスは、オプションの Derek Nexus モジュールをサポートするためにのみ必要です。オプションのモデルサーバーも装着して、ADME QSAR モデル、Auto-Modeller で作られたモデル、カスタムモデルを動作させることができます。StarDrop サーバーは 64 ビット Linux オペレーティングシステム (Red Hat Enterprise Linux 9/10 および Ubuntu 24.04 LTS) 上で動作します。
トライアル
デモ版お申込み
製品価格
※ 製品価格は、下記のお見積りフォームよりお問合せください。
- アカデミックライセンス
学位を授与する教育機関 (大学、大学院) には割引価格が適用されます。学位授与機関、および、そこに所属し教育・研究目的でご購入される方が対象となります。購入の際は、所属機関の証明書のコピーもしくは「ユーザー情報確認書」 (PDF / Excel) の送付が必要です。
お見積り・ご購入
サポート
インストール手順
StarDrop 8.x
StarDrop Derek Nexus
技術情報
モデルのダウンロード
Customer hub:各種モデルデータやアドオンデータ等が掲載されています。
- よくダウンロードされるモデル
P-糖タンパク質トランスポーター Model
P-糖タンパク質 (P-gp) は、MDR1 遺伝子によりコードされた ATP 駆動薬剤排出ポンプであり、薬の異なるクラスを含む広範囲の化学構造を輸送することが可能です。
Caco-2 Model
ヒト上皮結腸直腸腺がん細胞の Caco-2 ラインの単層透過性は、ヒト腸を介して吸収される化合物の活性を評価するために利用される一般的なインビトロモデルです。
導入事例
トップバイオ医薬品企業での実績
世界中の製薬企業における数千人の創薬化学研究者、トップバイオ医薬品企業の50%を含む専門家が、ヒット化合物から候補化合物への時間短縮のためにStarDropを活用しています。











ユーザーからのコメント
私たちはStarDropを日常的に使用し、複雑なマルチパラメータデータセットを可視化し、医薬品化学設計の次の段階に役立てています。ユーザーフレンドリーなインターフェースにより、メディシナルケミストはデータ分析と特性予測に容易にアクセスできます。まさに優れた創薬ツールです!
Andrew Stott
Director, Cerevance
StarDropでは一度に数千の化合物を扱っていますが、そのスピードとパフォーマンスはまさに驚異的です。かつては数千ページにも及ぶ特許出願書類を手作業で精査する必要がありましたが、今ではわずか10分、数回のクリックで完了します。ファイルをダウンロードし、StarDropで開き、R基分析を実行するだけで完了です。
Uwe Heinelt
Medicinal Chemist, Sanofi
StarDropは、メディシナルケミストにとって素晴らしいツールです。初期段階の医薬品化学に携わる場合でも、膨大な量のデータを扱う場合でも、非常に高速です。5分以内にSAR解析結果を表示し、将来の設計戦略に役立てることができます。
Sébastien Alazet
Medicinal Chemist, adMare Bioinnovations
ユーザー事例/活用提案
- StarDropの予測モデルによる抗炎症剤の改良
- 高速SAR解析でカナダの化学者を支援
- どのADMET特性を予測することが重要か?
- 創薬予測モデルを最大限に活用するには?
- 毒性予測のための Derek Nexus – どのパッケージが適切?
- 最も優れた代謝物予測ソフトウェアはどれですか?
- 創薬ソフトウェアにおけるデータ統合