 |
| サイトマップ | |
|
 |
| サイトマップ | |
|
 |
 |
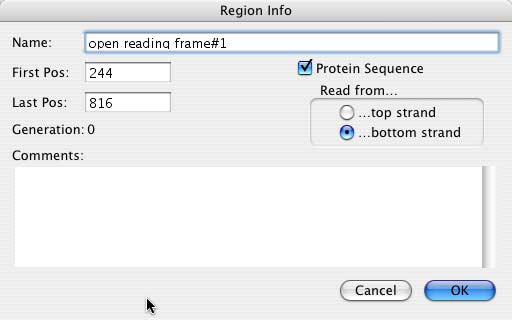 |
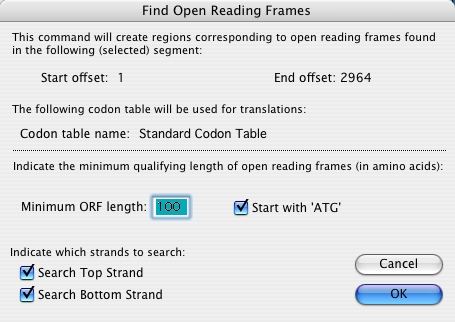
beta-galactosidase" と入力します。Comments ボックスに、コメントを付けることもできます。どちらのテキストも、検索に使用することができます。OK ボタンを押すとダイアログが閉じます。ampicillin resistance" に変更し、"beta-lactamase" のコメントを付けて、OK ボタンを押します。 |