 |
| サイトマップ | |
|
 |
| サイトマップ | |
|
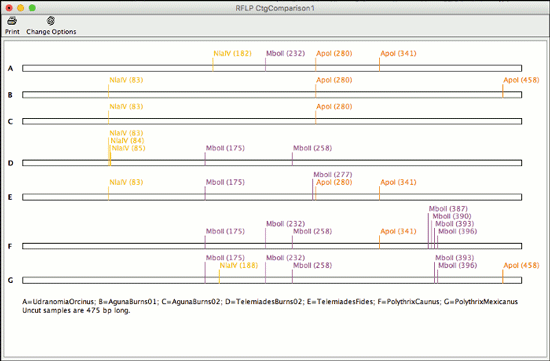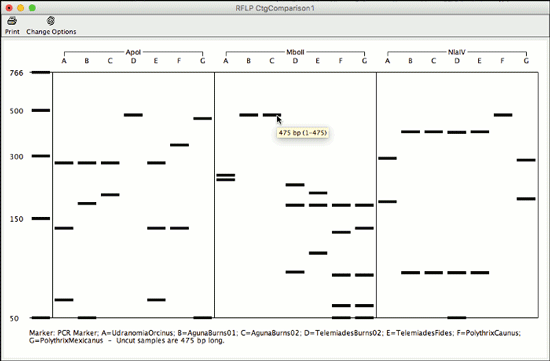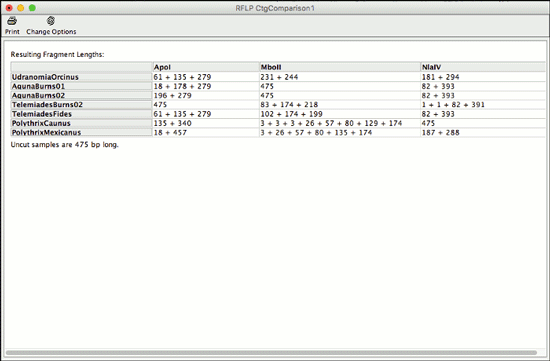
CodonCode Aligner 7.0 では、RFLP 解析が可能です。
この新しい機能により、コンティグにおけるすべてのサンプルについての切断位置の比較や RFLP 解析に用いた酵素の同定が可能です。また、期待する結果の仮想的なゲルを生成や、実際のゲルのレーンを確認するために印刷ができます。
さらに、シーケンスに対して異なる断片を生成する酵素を自動的に選択します。酵素を変更することができ、すでに生成された結果についてのオプションの表示や、異なる結果を横に並べて比較するために同じコンティグに対する新しい解析を開始することが可能です。
 |
CodonCode Aligner 7.0 では、NGS シーケンスデータについて新しく組み込みのアセンブリアルゴリズムが追加されました。SparseAssembler (バージョン6.0 で導入、現在もサポート) による NGS アセンブリと比較すると、新しくビルトインされた NGS のアルゴリズムは非常に長いコンティグや N50 を与えることができます。下表は、GAGE および GAGE-B のテストセットによるバクテリアゲノムデータを用いて、その結果を示しました。
| Data set | Read length | SparseAssembler N50 | CodonCode N50 | SparseAssembler largest contig | CodonCode largest contig |
|---|---|---|---|---|---|
| Staph. aureus | 101 | 28,877 | 37,787 | 74,595 | 93,238 |
| Vibrio cholerae | 250 | 2,726 | 80,570 | 19,705 | 207,445 |
アルゴリズムによる実際の結果の違いは、データセットによって変化する点にご留意ください。Vibrio データセットの 250 塩基のように、読み込みがより長い場合には、SparseAssembler と比較して新しくビルトインされたアルゴリズムでは、コンティグサイズと N50 値が 10 フォールド以上高くなることができます。
| ※ CodonCode Aligner に実装されている NGS アセンブリは現在のところ、バクテリアゲノムのような、小さなサイズのプロジェクトに制限されています。この制限は今後改良する予定です。 |