
DNA 配列のアセンブリとアラインメント
CodonCode Aligner は、DNA バーコーディング、シーケンスのアセンブリやコンティグの編集、 突然変異の検出に役立つ Windows および macOS 用プログラムです。親しみやすく分かり易いユーザーインターフェースを備えながら、Phred-Phrap と互換性のあるシーケンスのクオリティスコアをフルサポートする能力も備えています。
機能一覧
CodonCode Aligner を使ってできること
- テキストファイルからのシーケンスの読込み
- ABI ファイル、SCF ファイル、FASTQ ファイルからのシーケンスの読み込み
- End Clip 機能 (低品質のシーケンス末端を切り取る機能)
- ベクターのトリミング
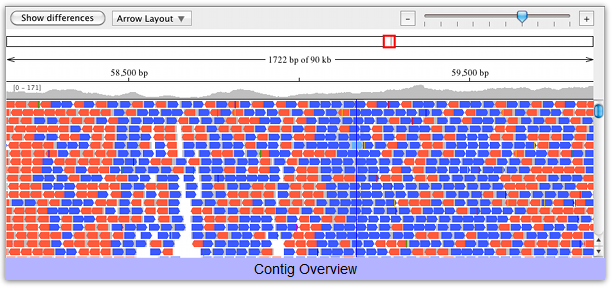
- コンティグのアセンブル
- 参照シーケンスへの整列
- 各コンティグのトレースへのリンクは維持しながら整列
- サンプル名に基づくグループ単位のアセンブルや整列
- ヘテロ接合型突然変異の検索
- ヘテロ接合性の挿入 (insertions) と欠失 (deletions) の分析
- プライマーの設計
- ユーザー定義した特徴 (features) 間のナビゲーション機能
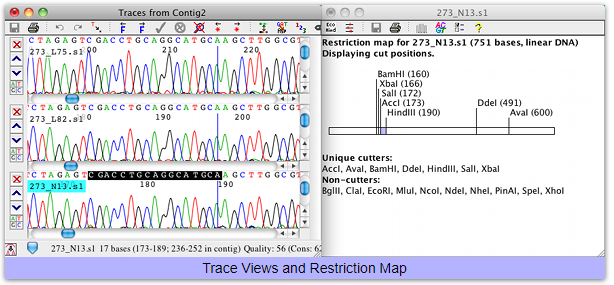
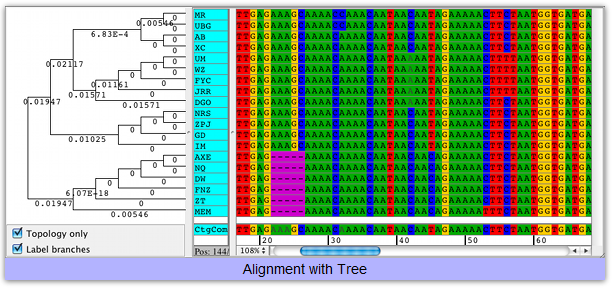
- 系統樹と制限マップの生成
- Aligner プロジェクトから直接 Phred および Phrap を実行
- シーケンスと特徴 (features) の出力
- 差分表とタンパク質翻訳を参照
- RFLP 分析用の酵素を検索し、仮想ゲルを作成
- メチル化されたシーケンスを分析し、生のトレースデータを表示
- 制限クローニングステップの計画と可視化
- 対立遺伝子を分離
- 関心領域 (features) の定義:低品質のコンセンサス領域など
- 細菌ゲノムと小規模な NGS シーケンスプロジェクトの組立て
- バーチャル制限クローニングを用いたクローニング実験の計画
- ギブソンアセンブリを用いたバーチャルマルチフラグメント
クローニング
CodonCode Aligner を使えば、難しいコマンドラインオプションや Perl プログラミングを学ばなくても、ベースコーリング (塩基判定) やシーケンスアセンブリのための最新アルゴリズムをフルに活用することができます。
インポート/エクスポート
- ABI、AB1、SCF フォーマットのシーケンスクロマトグラフのインポート。FASTA、FASTQ、Sam、GenBank、EMBL フォーマットのテキストファイルのインポート
- サンプルのサブセットのインポート (例:n 番目ごとのサンプル、プロジェクト内の選択したシーケンスに一致するサンプルのみ等)
- 新しいテキストシーケンスを作成し、アクセッション番号によって GenBank からインポート
- シーケンス、コンセンサスシーケンス、アラインメント、プロテイントランスレーションを SCF、FASTA、FASTQ、NEXUS/PAUP、Phylip にエクスポート
シーケンス編集とトリミング
- シーケンスとコンティグのマニュアル編集:サンプルやコンティグエンドの削除、コンティグ分割など
- 自動化された編集ツール: 曖昧さの呼び出しと削除、コンセンサスの一致、低品質の塩基を N に変換、自動編集のアンドゥ
- ツールバーボタンやキーボードショートカットを使った素早い操作:ユーザーが興味のある領域を定義可能
- 低品質のシーケンスを削除するためのエンドクリップ:ベクトルコンタミネーションを削除
シーケンスアセンブリ
- ローカル、エンド・トゥ・エンド、ラージギャップ (cDNA to Genomic) アルゴリズムを使ったフラグメントのアセンブル
- 正確で、品質ベースのコンセンサスシーケンスがマニュアル編集の必要性を最小化
- 名前でアセンブルし、多数のクローンや標本に対して別々のコンティグを作成
- Phrap でアセンブル
シーケンスアラインメント
- リファレンスシーケンスへのアラインメント:cDNA を Genomic DNA にアラインメント
- Bowtie2 アライメントを作成し、ペアエンド読み取りを表示
- 「コンティグのコンティグ」を構築:コンティグを MUSCLE や Clustal で相互にアラインメント
- ダブルクリックでコンティグのアラインメントからオリジナルのトレースに移動
- データの一貫性を維持しながら、シーケンストレース、コンティグ表示、コンティグのアラインメントを編集可能
シーケンストランスレーション
- すべてのシーケンスに対するプロテイントランスレーションを表示
- アミノ酸と塩基の表示を切り替え可能
- トランスレーションベースの背景色を表示
- 注釈つきのコーディング領域に、コンセンサスシーケンスのプロテイントランスレーションを表示
バーチャルクローニング
- 制限クローニング操作のシミュレーション、ベクターとインサートにマッチするオーバーハングを簡単に検出可能
- 最大 15 フラグメントのバーチャルギブソンアセンブリ
- 平滑末端 PCR および TOPO クローニング
- 指向性 TOPO クローニング
- TA クローニング
- クローニングプライマーを自動的に選択
- 手動でプライマーを編集して、スペーサー等を挿入またはリーディングフレームを調整
- 実験中にプライマーを直接エクスポート
- リーディングフレームによるクローンの検証
制限マッピング
- 制限酵素の任意の組み合わせを使った制限マッピングを構築
- 生成者の認識サイトの大きさ、オーバーハング、カットフリクエンシーによって選択
- シングルラインマップ、マルチラインマップ、テキスト、または仮想ゲルとして表示
- コピー時に制限マップ上をクリックして制限フラグメントを選択
RFLP 分析
- 異なるゲルレーンを生成する制限酵素を自動的に識別
- コンティグ内のサンプルのカットサイトを比較
- 予想される仮想ゲル、各酵素のフラグメントの長さ、または切断されたサイトの位置合わせされたサンプルを表示/印刷
プライマー設計
- PCR およびシーケンシングプライマーの設計
- クローニングのため固定位置に PCR プライマーを作成
- 認識しやすいように、テンプレートシーケンスでプライマーを強調表示
- プライマーごとに、詳細なプライマー情報とともにプロジェクトにインポート可能
- プライマーを順番どおりにエクスポート
系統樹
- コンティグに対して近接結合 (Neighbor-Joining) 樹を作成
- 選択した塩基に対してツリー (樹) を作成し、特定の領域の差によってのみサンプルをソート
- ツリーによってコンティグを分割
突然変異の検出と分析
- シーケンストレースにおいて二次ピークを呼び出し
- アセンブルまたはアラインメントされたコンティグ内のヘテロ接合 SNP の高感度検出
- 突然変異のアミノ酸レベルの影響を表示/エクスポート
- ヘテロ結合の挿入と削除を正確かつ高角度で検出
- ヘテロ結合インデルを、より短い/より長い疑似対立遺伝子 (pseudoallele) に分割
- ワイルドタイプトレースを除去して、突然変異した対立遺伝子を明示
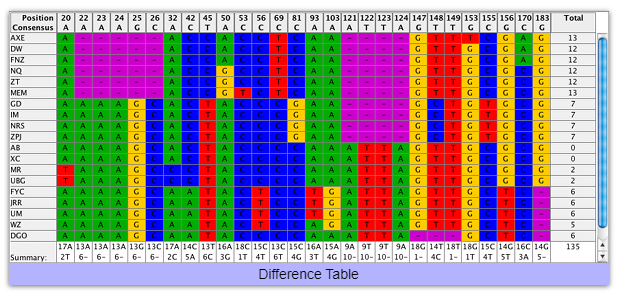
差分表
- 差分表を使ってコンティグ内の差を特定
- 差分表内のセルをクリックして差を確認し、コンティグ内の差分をナビゲート
- 差分表の簡略版で、すべての差分の概要を把握
- フィルタを使って興味のある部分の差分だけを参照可能
- 差分をエクスポート
カスタマイズ
- ヌクレオチド、シーケンス品質、アミノ酸によって塩基を色付け
- コンセンサスシーケンスに一致する塩基を非表示化
- ツールバーのボタンとボタンのスタイルを選択可能
- プリファレンス画面で詳細設定が可能
- プリファレンスファイルをロック、保存、読み込み
- スクリプトを使って、繰り返しの作業を自動化
メチル化分析
- 亜硫酸水素塩修飾後のシトシン、PCR、ABI データからのキャピラリーシーケンスのメチル化の分析
- 「生の」ABI トレースデータの表示
- メチル化分析では生のトレースデータを使用するため、トレースの「正規化」によるアーティファクトを回避
- 個々のシーケンスのメチル化結果と、コンティグ塩基の観測範囲を取得
アップデート情報
各アップデート情報は こちら をご確認ください
動作環境
CodonCode Aligner 12.0 動作環境
Windows
- Windows 10 以降 推奨; Windows 11 対応
Mac
- Mac OS X 10.10 以降; Sequoia (15) 対応
ライセンス
ライセンス
- 一般用
企業主体の研究機関、事業組合等が対象です。
Phred、Phrap、Phred-Phrap パッケージのコンポーネントは含まれていません。 - 教育/官公庁用
大学、非営利研究機関が対象です。
Phred、Phrap のワークステーションバージョンが無償で付属しています。 - 学生用
2 年間の期間限定ライセンスです。ご購入時に、エンドユーザーの学生証のコピーが必要です。他のユーザーとの共用はできません。ライセンスの有効期間中であれば、有償で教育用ライセンスにアップグレードできます。
年間保守
一般用および教育/官公庁用ライセンスの新規購入価格には、1 年間の保守 (フリーアップグレード、技術サポート、ライセンスキー再発行) が含まれています。保守の更新は任意です。保守が失効した後に最新版を入手するにはアップグレードの購入が必要です。
Single User ライセンス
- インストール可能台数
1 ライセンスあたり 1 台 - ライセンスキー
ライセンスキーはハードウェア固有です。
CodonCode Aligner をインストールした PC の「computer ID」を CodonCode Corporation にメールで送信すると、ライセンスキーが発行されます。
同時使用 (ネットワーク) ライセンス
Aligner License Server プログラムによって、同じネットワーク上にある CodonCode Aligner を管理します。 CodonCode Aligner を実行したい端末から、ネットワーク経由でライセンスサーバーにアクセスし、ライセンスキーをチェックアウトします。出張などの場合は、期限付きキーをチェックアウトする (オフライン用にキーを払い出す) ことも可能です。
- インストール可能台数
Aligner License Server:1 ライセンスあたり 1 台
CodonCode Aligner:無制限 (一度にアクセスできる端末数は、購入したライセンス数まで) - ライセンスキー
サーバー固定のライセンスキーが必要です。
Aligner License Server をインストールしたサーバーの「computer ID」を CodonCode Corporation にメールで送信すると、ライセンスキーが発行されます。
ライセンスキーの交換
CodonCode Aligner または Aligner License Server をインストールしたマシンを変更する場合は、ライセンスキーの交換が必要です。 新しいライセンスキーが発行された後に、元のライセンスキーを使用すると、片方または両方のキーが無効となります。
ライセンスキーの交換は、年間保守の契約期間中、および年間保守の失効から最大 2 年間は無償です。年間保守が失効して 3 年以上経過している場合は有償となります。
トライアル
デモ版お申込み
- デモ版の有効期間は 30 日間です。
- 製品版と同じ機能をご利用いただけます。
製品価格
製品価格
| 対象 | 製品名 | 税込価格 |
|---|---|---|
| 一般用 | CodonCode Aligner 12 Single User | ¥ 355,300 |
| 一般用 | CodonCode Aligner 12 同時使用1 | ¥ 355,300 |
| 教育/官公庁用 | CodonCode Aligner 12 Single User 教育/官公庁用 | ¥ 177,100 |
| 教育/官公庁用 | CodonCode Aligner 12 同時使用1 教育/官公庁用 | ¥ 177,100 |
| 学生用 | CodonCode Aligner 12 Single User 学生用 (2年間) | ¥ 79,200 |
アップグレード
| 対象 | 製品名 | 税込価格 |
|---|---|---|
| 一般用 | CodonCode Aligner 12 Single User アップグレード | ¥ 178,200 |
| 一般用 | CodonCode Aligner 12 同時使用1 アップグレード | ¥ 178,200 |
| 教育/官公庁用 | CodonCode Aligner 12 Single User 教育/官公庁用 アップグレード | ¥ 89,100 |
| 教育/官公庁用 | CodonCode Aligner 12 同時使用1 教育/官公庁用 アップグレード | ¥ 89,100 |
アップグレードには 1 年間の保守 (フリーアップグレード、技術サポート、ライセンスキー再発行)が含まれます。
お見積り・ご購入
※アップグレードについては、現在お持ちの製品名、バージョン、ライセンス番号 (Computer ID) を必ずご記入ください。
サポート
チュートリアル
- CodonCode Aligner チュートリアル一覧
CodonCode Aligner 入門(日本語訳)、および、開発元が提供しているチュートリアル (英語) にアクセスできます。
インストールおよびライセンスキーの入手方法
- シングルユーザーライセンス インストール手順 (PDF)
- 同時使用ライセンス インストール手順 (PDF)
- インストーラーのダウンロードはこちら (開発元サイト)
CodonCode Aligner (Mac/Win)
Aligner License Server (Mac/Win)
| ※ ライセンスキーはハードウェア固有です。インストールしたマシンを変更する場合は、ライセンスキーの交換が必要です。 新しいライセンスキーが発行された後に、元のライセンスキーを使用すると、片方または両方のキーが無効となります。 ※ ライセンスキーの交換は、年間保守の契約期間中、および年間保守の失効から最大 2 年間は無償です。年間保守が失効して 3 年以上経過している場合は有償となります。保守が切れていて、ライセンスキーの交換をご希望の場合はお問い合わせください。 |
サポートお問い合わせ
導入事例
新型コロナウイルスの分析プロセスで CodonCode Aligner を使った論文の例
- Emergence of two distinct variants of SARS-CoV-2 and an explosive second wave of COVID-19: the experience of a tertiary care hospital in Pune, India / 9 Jan, 2022, / By Shubham Shrivastava et al. / Archives of Virology
(SARS-CoV-2 の 2 つの異なる亜種の出現と COVID-19 の爆発的な第 2 波:インド、プネの 3 次医療病院での経験から)
詳細は こちら(SpringerLink のサイトへ) をご覧ください。 - A Sanger-based approach for scaling up screening of SARS-CoV-2 variants of interest and concern / 25 Mar, 2021, / By Matheus Filgueira Bezerra et al. / The Oswaldo Cruz Foundation (Fiocruz)
(懸念される SARS-CoV-2 変異体のスクリーニングを拡大するための Sanger ベースのアプローチ)
詳細は こちら(Fiocruz のサイトへ) をご覧ください。 - A Half-Day Genome Sequencing Protocol for Middle East Respiratory Syndrome Coronavirus / 19 Feb, 2021, / By Kwan Woo Kim et al. / Frontiers in Microbiology (Frontiers Media S.A.)
(中東呼吸器症候群コロナウイルスの半日ゲノム・シーケンシング・プロトコル)
詳細は こちら(Frontiers in Microbiology のサイトへ) をご覧ください。 - Maturation and persistence of the anti-SARS-CoV-2 memory B cell response / 2 Feb, 2021, / By Aurélien Sokal et al. / National Center for Biotechnology Information, U.S. National Library of Medicine
(抗 SARS-CoV-2 メモリー B 細胞反応の成熟と持続性)
詳細は こちら(NCBI のサイトへ) をご覧ください。

Jack Pettigrew 教授と彼の研究グループは、オーストラリアの古代のロックアートの年代特定のために CodonCode Aligner を使用しています。最近の発見では Bradshaw ロックアートは、岩絵と密接に結合するさまざまな微生物を含むバイオフィルムで覆われていることがわかりました。彼らは、これらの微生物が持つ進化情報を使って、岩絵の年代を特定したいと考えています。

パキスタンの国立バイオテクノロジー遺伝子工学研究所(the National Institute for Biotechnology and Genetic Engineering)と、カナダ・オンタリオ州生物多様性研究所(the Biodiversity Institute of Ontario)の共同プロジェクトである、パキスタンの経済的に重要な昆虫種のバーコードシーケンス・データの分析における CodonCode Aligner の使用例です。

このユーザー事例では、オオミチバシリ(学名 Geococcyx californianus)の個体数と保全遺伝学に関して、オオミチバシリの遺伝的変異と遺伝子流動の評価に、ミトコンドリアと核マーカーをどのように使用しているかをご紹介します。

ユネスコ世界遺産であるアルダブラ環礁では、固有の landbird 2種 (アルダブラベニノジコ と アルダブラクイナ) の分類学的地位と、外来種のベニノジコと固有種のアルダブラベニノジコの交配による影響を研究するために、CodonCode Aligner が使用されています。

ヨーロッパアワノメイガ (ECB) の進化、種分化と突然変異のさまざまな側面における研究事例です。ECB の休眠後の発育と個体群間の遺伝子流動との関係や、時間的隔離と遺伝子座との関連性にも注目した研究です。

このユーザー事例では、CodonCode Aligner を使って、電気ナイフフィッシュ Eigenmannia の反復 DNA 要素の動態、保全、分散と進化について考察しています。
