 |
| サイトマップ | |
|
 |
| サイトマップ | |
|
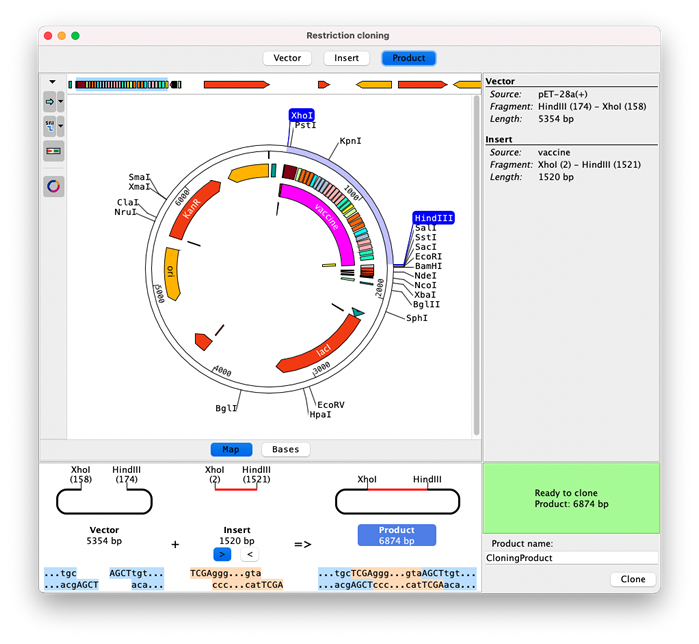
CodonCode Aligner 10 では、仮想的な制限クローニングを実行できます。
|
線形または円形のマップ、又は、塩基レベルとしてベクターと挿入シーケンスを表示できます。制限酵素やフィーチャー (注釈) をクリックすることで酵素とクローンするセクションを容易に選択できます。
画面下の部分には、ベクターと挿入シーケンスがどのように組み合わされているか、切断部位に基づいて生成物ができるかが表示されます。
画面左側にあるツールバーボタンには、どのマップ、フィーチャー、および、制限酵素を表示するかを変更するためのオプションが用意されています。例えば、一度切断した制限酵素だけを表示するという具合です。
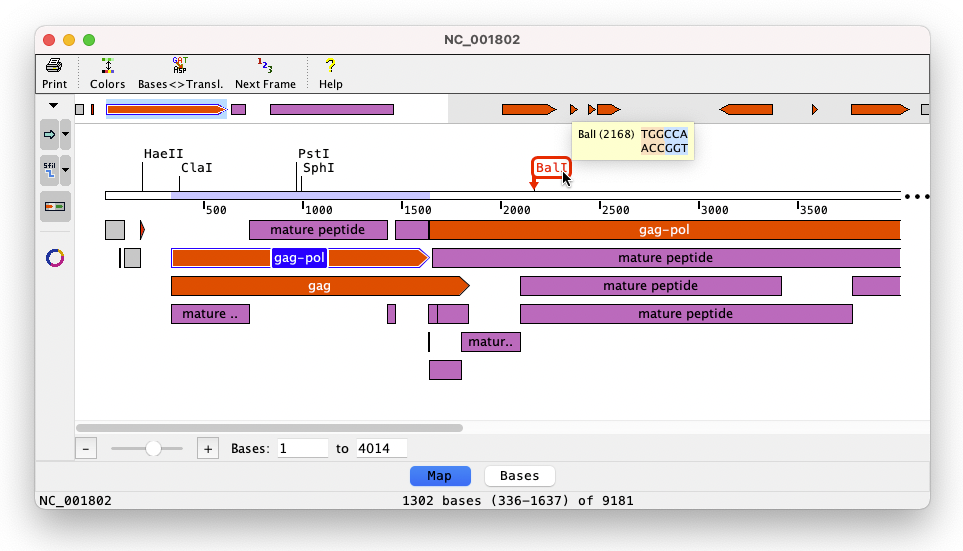
Aligner 10 には、クローニング機能と連携したシーケンスマップを表示できる機能が追加されました。円形と線形のマップを表示したら、そこにカスタマイズ可能なフィーチャーと酵素切断部位が表示されます。
|
画面上部にミニマップとして概要が表示されます。シーケンス内の特定の領域を選択するには、そのフィーチャーをクリックするか、Shift キーをおしながら2つの酵素をクリックします。マウスオーバーすると、切断部位とフィーチャーに関する詳しい情報が表示されます。また、画面下のズームオプションを使用すれば、特定の領域をズームすることができます。特定の領域を選択したり移動したあとにマップ表示と塩基表示を素早く切り替えることができます。また、画面左にあるボタンを使えば、表示するフィーチャーや酵素といったマップオプションをカスタマイズできます。
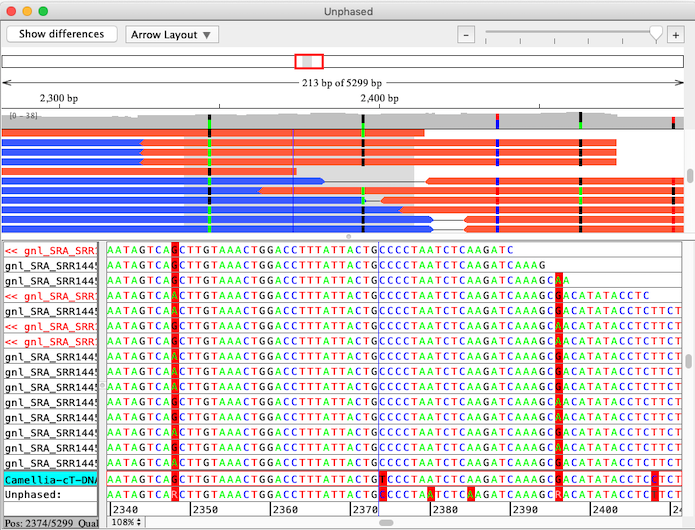
CodonCode Aligner の “Separate Alleles” は、小規模な次世代シーケンスプロジェクトにおいて二倍体生物由来の対立遺伝子間の連鎖を特定するための機能です。対をなす末端配列の中の連鎖する対立遺伝子をあらわした事例を以下に示します:
|
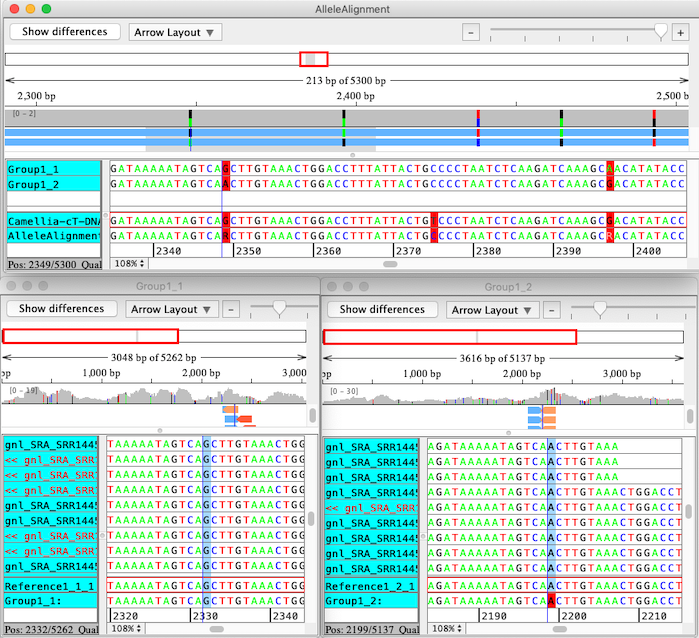
CodonCode Aligner は、複数ステップのプロセスを経て各配列を参照配列にアラインメントし、対立遺伝子の連鎖を識別し、配列対を連鎖グループとして取り出します。この分析の最終結果は、連鎖した対立遺伝子をグループに分けた個々のコンティグです。これらは、CodonCode Aligner の “Compare contigs” 機能を使ってオリジナルの参照配列にアラインメントされます。以下のスクリーンショットは、このような分析を行った結果を示します。画面上には、”contig of contigs” の対立遺伝子のアライメントが、画面下には、対立遺伝子に固有のコンティグが表示されています。
|