チュートリアル 20:Find Sequence
- GCK を起動します。
- File > Open… を選択して Tutorial Files フォルダにある pBR322 という名称のファイルを開きます。
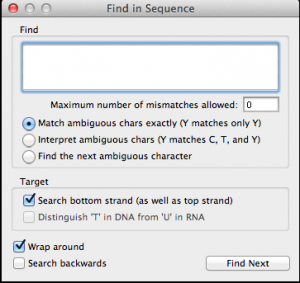
- Edit > Find.. . を選択して図 2.91 に示すダイアログを開きます。
図 2.91 Find Sequence ダイアログ
 |
- Find ウィンドウに、以下に示すヌクレオチド文字列から先頭の何文字かを入力 (またはコピー&ペースト) します。
ACTCTTCCTTTTTCAATATTATTGAAGCATTTATCAGGGTTATTGTCTCATGA
GCGGATACATATTTGAATGTATTTAGAAAAATAAACAAATAGGGGTTCCGCG
注意:上記の塩基配列は、アンピシリン耐性 (Ampicillin Resistance) 遺伝子のプロモーター領域をあらわします。
- Find Sequence で使用する検索ダイアログは、大文字と小文字を区別するので、検索プロセスを精密に制御できます。検索文字列として大文字で入力されたヌクレオチドは、ターゲットのシーケンス (保存領域) と厳密に一致します。一方、小文字で入力されたヌクレオチドは、塩基配列がミスマッチを許容する箇所に利用することができ、その文字列を許容するミスマッチの最大数はクエリーで定義することができます。
検索文字列には、あいまい文字列 (N, Y, W, B, D, など) も利用できます。これは標的配列のヌクレオチドにばらつきがある場合に便利です。利用できる Legal Nucleotide Characters の一覧表は、GCK マニュアルの Appendix をご覧ください。
チェックボックスを使えば、検索対象を上鎖 (Top strand) のみか上下両鎖 (Top および Bottom Strand) か、また、検索が一致するたびに塩基配列の先頭に戻るか否かを定義することができます。これにより起点 (origin) がスパンする環状コンストラクトの塩基配列を見つけることができます。
- 実行する追加のパラメータ設定が終わったら、Find Next ボタンをクリックして検索を開始します。
- 検索結果として範囲 4156 – 4260 のヌクレオチドが見つかり、コンストラクト内にハイライトで表示されるはずです。
以上でチュートリアルは終了です。pBR322 ファイルを閉じてください。なお、他の人がこのサンプルファイルを使うかもしれませんので、ここで加えた変更内容は保存しないようご注意ください。


