 |
| サイトマップ | |
|
 |
| サイトマップ | |
|
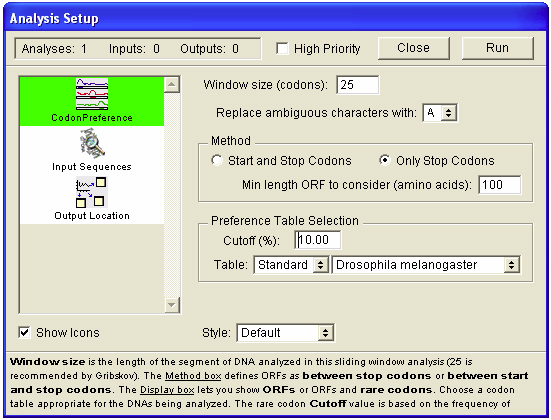
この解析は、DNA のコーディング領域を見つけるのに使用します。Gribskov, M. らの論文 [Nucl. Acids Res. 12(1):539 (1984)] に依拠します。生成されるコドン選好プロット (codon preference plot) は、遺伝子やエクソンの同定や、DNA シーケンスの挿入や欠失によって生ずるエラー (突然変異) を検出するのに役立ちます。図 4.19 に示すのはこの解析で使うセットアップパネルです。
 |
この解析では、調査する生物種が使用するコドンを定義したコドン頻度テーブル (codon frequency tables) h を使用します。
| ※ h. 生物種の多くは同じ遺伝コードを使用しますが、コドン選好テーブルには大きな違いがある点に注意してください。翻訳テーブルには各コドンでコードされるアミノ酸に関する情報が含まれますが、コドン選好テーブルにはコドンの使用に関する情報が含まれます。 |
コドン選好テーブルは、各コドンとそれらが特定のアミノ酸を指定するのに使用される頻度をまとめたテーブルです。例えば、グリシン (glycine) に対応するコドンは4つありますが、それらの使用率はどの生物でも等しく 25% になる訳ではありません。これら4つのコドンは生物によってそれぞれ異なる頻度で使用されます。利用可能なコドン頻度テーブルは、解析セットアップパネルの下にあるポップアップメニューから選択することができます。このポップアップには、Gene Inspector で提供される 48 種類の標準 (Standard) テーブルと、ユーザー独自に定義できる User テーブルが用意されています (詳細については、チュートリアル 20: オリジナル解析テーブルの作成と、“Editing Translation and Codon Preference Tables” をご覧ください)。GI Data フォルダの User Table フォルダにある User テーブルならタイプが正しければいずれもこのポップアップメニューで選択することができます。リストに目的の生物が無い場合は、関連する生物由来のコドン選好テーブルを使用することもできますが、目的の生物のコドン頻度テーブルが選択した生物種のものと類似しているという保証はありません。リストに目的の生物が無い場合は、コドン選好テーブルに依存しない TestCode 解析 ("TestCode") を試してみるか、お持ちのコーディングデータを使用してコドン選好テーブルを新たに作成してください。
この解析は、特定コドンの認識とそれに対応するテーブル内の値をルックアップする能力に依存するため、シーケンス内に不明な文字があった場合、出力結果の有意性に問題が生じることもあります。曖昧文字の取り扱いについては、“TestCode” で説明してあります。その内容は、ここで説明する CodonPreference 解析とも関連があります。曖昧な文字をこの解析でどのように取り扱うかは、図 4.19 に示すセットアップパネルを使って指定することができます。
デフォルト (標準) で指定された窓サイズ 25 コドンは、著者 (Gribskov, M. ら) の推奨する値です。これは、コドン使用を調査する DNA セグメントのサイズをあらわします。この移動窓におけるコドンの使用数が、目的の生物種のコドン頻度テーブルの度数と比較されることになります。実際の使用頻度がテーブルの値に近ければ近いほどプロットの値は高くなります。図 4.20 に示すのは、CodonPreference 解析の出力結果です。読み枠 (reading frame) ごとに1つの曲線が描画されています。カットオフ・ラインより上の値はいずれも 95% の信頼水準でコーディング領域になることをあらわします。この事例の場合、読み枠3のおよそ 1600 ~ 3700 nts の範囲に良好なコーディング領域を確認することができます。
CodonPreference プロットには、別なオプションを表示させることもできます。
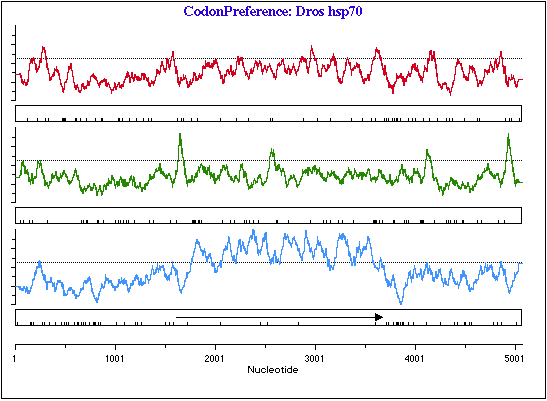 |
コドンの中には、ある生物ではめったに使用されないものもあります (同義語コドンの任意のセットで通常使用される回数の 10% 以下)。このようなコドンは、実際のコーディング領域で見つかる見込みはほとんどありません。レアコドンの出現をプロット長に沿ってプロットすることで、多くの場合コーディング領域になりそうな部分を判定することができます。読み枠3の実際のコーディング領域の内部では、レアコドンの頻度をあらわす目盛りが、他のプロットと比較して非常にまばらである点に注目しましょう。このことは、この領域が実際のコーディング領域であることを示す更なる裏づけとなります。レアコドンをプロットしたい場合は、セットアップパネルで指定することができます (図 4.19)。
最後に、プロットには実際の読み枠 (ORF) が矢印で描画されます。ORF は、いずれの終止コドンも含まれない DNA セグメントを表すものです。解析の出力で ORF が描画される最小の長さを図 4.19 に示すセットアップパネルを使って指定することができます。すべての ORF が必ず「開始」コドンで始まるか (原核生物ではその可能性が高い)、真核細胞遺伝子におけるイントロンの結果として生ずる、任意のコドンでの開始も許容するかを指定することもできます。ORF は、図 4.20 に示すように水平の矢印で表示されます。
ORF の矢印は、CodonPreference 出力オブジェクトがターゲット状態i であればマウスで選択することができます。ORF を選択状態にすると、関連する DNA やペプチド配列を新規シーケンスエディタのウィンドウに展開することができます。これに関しては、 “Open Reading Frames” と、チュートリアル 17: Testcode (インタラクティブな解析の紹介) で詳しく説明してあります。
| ※ i. オブジェクトがターゲット状態になっていない場合は、Option キーを押しながらマウスでクリックすることで ORF を選択状態にすることができます。Option キーをマウスと組み合わせて使うことでオブジェクトのパーツを選択することができます。 |
コーディング領域を同定する3つの手法が CodonPreference 解析の活用、および、レアコドンと読み枠 (ORF) の表示により、ひとつの出力オブジェクトとしてそれぞれ提供されます。図 4.20 に示す3つの情報が実際の Drosophila hsp70 のコーディング領域と一致している点にに注目してください。