 |
| サイトマップ | |
|
 |
| サイトマップ | |
|
シーケンスエディタは、シーケンスの多重整列 (multiple sequence alignments) の結果をあらわすウィンドウにもなります。シーケンスの多重整列は、通常の Analysis Setup ウィンドウを使って解析の一つとして作成するか、もしくは、シーケンスエディタ・ドキュメントの内部から直接作成することができます。詳細は、チュートリアル 5: シーケンスの多重整列をご覧ください。
現在シーケンスエディタウィンドウに開いている全てのシーケンスを整列するには、Sequence > Alignment > Align All Sequences... を選択します。これを選択すると、タンパク質の整列の場合は、図 3.10 に示すようなダイアログボックスが表示されます。この事例の場合は、入力できる多数のパラメータが用意されています。Gene Inspector が使用する多重整列アルゴリズムは、Clustal V と呼ばれるものです [Higgins, D.G., A.J. Bleasby, and R. Fuchs, Comp. Appl. Biol. Sci. 8(2):189 (1992)]。多重整列の実行コードは、EMBL (欧州分子生物学研究所) の Dr. Des Higgins 氏のご厚意によるものです。
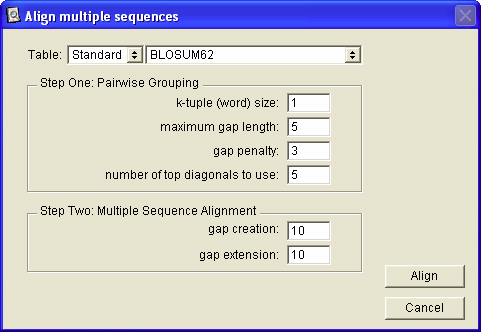 |
多重整列を成功させるには、最も類似するシーケンスがどれとどれがであるかを知る必要があります。これは、「ガイド」となる大まかなツリーを計算して漸進的 (progressively) に行っていきます。このガイドツリーは多重整列の計算中、徐々に大きなグループを整列していくためのガイドとして使用されます。互いに類似するシーケンスの様々な整列結果を並べ変えることで、最終的なシーケンスの多重整列が完成します。
まずはじめに、様々なシーケンスを比較するために使用するテーブルを選択する必要があります。これは、他の解析でテーブルを選択するのと同じやり方でポップアップの Table メニューを使って行います。
これから行うことに熟知していなかったり、色々と実験することに関心がない場合は、パネル内のその他のパラメータについてはデフォルト値のままにしておくのが良いでしょう。パラメータを不用意に変更すると、誤解を招くおかしな結果になる場合がありますので、とりわけステップ1のパラメータについては注意して取り扱うようにしてください。ステップ2のパラメータを変更すると、整列の結果が変わり、異なる整列スコアになります。以下に示すのは、各パラメータの内容を Clustal V コードに添付されたドキュメントに基づいて簡単に説明したものです。