 |
| サイトマップ | |
|
 |
| サイトマップ | |
|
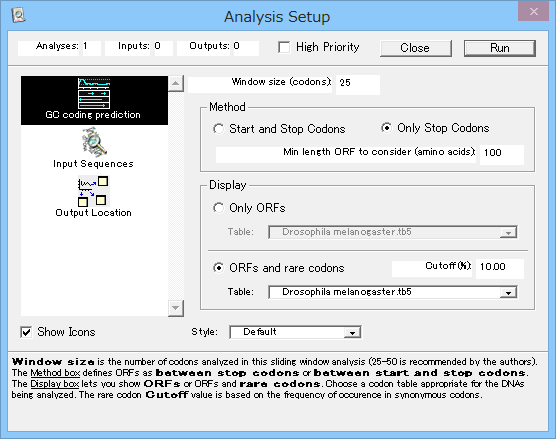
GC Analysis では、Bibb らの論文 [Gene 30:157 (1984)] で発表されたアルゴリズムを使用します。この解析は、生物のコドン使用に関して同義語コドンの中に選好するものが存在するという事実を利用します (詳細は "CodonPreference" 参照)。この偏りの結果、特定の読み枠の三番目のコドンの位置に、G+C 成分の極端な非対称性が多く存在します。図 4.33 に示すのは、GC Analysis のセットアップパネルです。
 |
 |
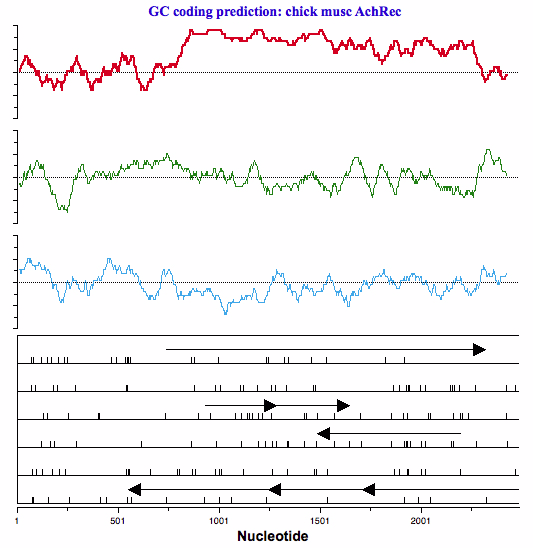
この解析の出力結果には、図 4.34 に示すように DNA の様々な位置の G+C 成分の分布が表示されます。最初の曲線は、位置1 (読み枠 #1) で開始する全ての3番目のヌクレオチドの G+C 成分を表し、2番目の曲線は、位置2 (読み枠 #2) で開始する全ての3番目のヌクレオチドの G+C 成分を表し、3番目の曲線は、位置3 (読み枠 #3) で開始するものを表します。この事例の場合、読み枠1には読み枠 (ORF) 750~2700 に対応するプラトー (高い値で推移する部分) が明確に存在します。GC Analysis には、単一の読み枠の特異性を調べる能力があるので、読み枠のフレームシフトの原因となる配列決定の誤り (sequencing errors) を同定するのに利用することができます。コーディング領域における配列決定の誤りは、読み枠のずれになる可能性があります。読み枠のずれは、2つに分かれた上昇領域として GC Analysis の出力プロットに表示されます。
指定があれば、プロットの下に実際の読み枠とレアコドンの使用頻度が表示されます (図 4.33)。このプロット領域については、チュートリアル 17: Testcode (インタラクティブな解析の紹介) で説明してあります。
 |