| 本アーティクルは The Mathematica Journal (volume 17, 2015) で発表されたもので、著作権は Wolfram Research, Inc. に属します。 |
| "This article was previously published in The Mathematica Journal (volume 17, 2015) and the copyright holder is Wolfram Research, Inc." |
本アーティクルでは、例えば腫瘍性肝組織 対 肝硬変肝組織のような、2つの生物学的状態の間の表現型の違いを作成する、最も直接的に責任のある遺伝子を同定するために設計された計算生物学アルゴリズム RIFA の実装について説明します。
マイクロアレイ技術の発明によって、科学者は2つの生物学的状態間の遺伝子発現の全体的な変化を測定するための手段を持ちました [1]。これは各々の科学者のお気に入りの実験システム中の差異的発現遺伝子の長いリストを記述した何千もの科学出版物につながっています。差異的発現遺伝子のリストは、2つの表現型間の違い (表現型は 1つまたは複数の特性の物理的発現を表します) を理解する上で重要な最初のステップですが、表現型の変化を制御する最も直接的に責任のある遺伝子を特定するのに、しばしば十分でないということが生物学者に徐々に明らかになってきました。2つの生物学的状態間の差異的発現遺伝子は、それらの違いを説明する上で重要であるかもしれないことは事実ですが、発現が変わらない遺伝子も表現型の違いを動かす中心になる可能性があります。
生物学に不慣れな人にとっては、大まかな類推は有用かもしれません。製造ウィジェット (健全な肝臓組織などの特定の表現型) をゆだねられた管理者の責任の下に、管理者 ("支配者" 遺伝子) と従業員 ("奴隷" 遺伝子) がいる製造環境を考えてみます。健全な肝臓組織 (青ウィジェットを製造) から癌性肝臓組織 (赤ウィジェットを製造) への推移のような表現型の変化は、以下によって達成することができます: (1) 従業員が働く割合を変化させます (管理者が従業員をどなる場合などに発生します。これは差異的発現に類似します) ; および/または (2) 管理者が従業員に与えている指示を変更します (指示の量を一定に保ちますが、指示に含まれる情報を変更します。これは "支配者" 遺伝子の変化に類似しています) ;および/または (3) (1) と (2) のシナリオを組み合わせます。シナリオ (1) では従業員 ("奴隷" 遺伝子) が以前よりも速くまたは遅く作業を開始するため、表現型の推移があります。これは、誤った時間に多すぎる、または少なすぎる遺伝子生成物を生成し、異なる生成物 (赤ウィジェット表現型) が作られるような製造の全てに波及します。この状況では、従業員への管理者の指示は一定のまま (青ウィジェットを製造) ですが、より大きい ("叫び") もしくはより小さい ("ささやく") ボリュームで話されす。シナリオ (1) はマイクロアレイ実験で測定することができる情報の種類を反映します。その唯一の目的は2つの生物学的状態の間で表現型が変化する遺伝子を特定することです。シナリオ (2) では、従業員の仕事の割合は一定のままですが、管理者から受けている指示が変更されるため、異なる色のウィジェット (表現型) を製造します。表現型の違いを生成する主要な原動力は ("支配者" 遺伝子中の変化のような) 管理者から従業員 ("奴隷"遺伝子) への指示の変化であり、従業員の生産率の違いではないので、差異的発現遺伝子の長いリストを生成することによって、シナリオ (2) による表現型の違いはほとんど発見されません。
これらの理由から、計算生物学者はその遺伝子が差異的に表現されるかどうかにかかわらず、表現型の変化の動かすことに主に責任がある遺伝子をハイライトすることに優れているアルゴリズムを開発し始めました。これが、ここで提示する regulatory impact factor analysis (RIFA) アルゴリズムの目的であり、表現型の変化を動かすための最も直接的な責任のある遺伝子をハイライトすることです。RIFA は、2つの生物学的状態中の遺伝子間の結合の違いを検出するための、計算的に扱いやすい方法を提供します。図1は結合性の前提と2つの生物学的状態間の結合性の違いを示しています。
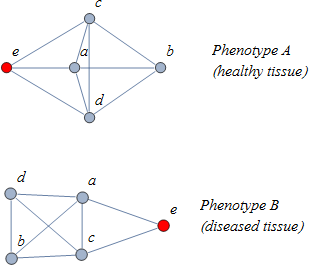
図1.2つの異なる生物学的状態 (表現型) の同じ5つの遺伝子からなる2つの遺伝子ネットワーク。各頂点は遺伝子を表し、各辺は遺伝子間の接続を表します。標準の差異的発現の研究では、各遺伝子の発現レベルは2つの生物学的状態の間でそれ自身と比較されますが、異なる遺伝子間の潜在的な関係を無視しています。カジュアルな観察者が上記の2つのネットワークを比較すると、それぞれのネットワークの形が異なることはすぐに気づきます。そして、違いはそれぞれの生物学的状態の中の遺伝子間の関連性の変化によって動かされます。
Regulatory impact factor analysis (RIFA) は、Hudson、Reverter、Dalrymple の独創的な研究に基づいており [2]、それは、差異的発現遺伝子の研究から得られる基本的な情報から計算される、3つの高次メトリックの全てを紹介します。これらのメトリックの目的は、差異的発現遺伝子 (上記の類推を用いた "奴隷" 遺伝子) と、転写因子 (上記の類推を用いた "支配者" 遺伝子) として知られる遺伝子発現調整遺伝子のグループの間の接続を定量化するために、遺伝子発現の研究にある情報を使うことです。3つのメトリックは次のようになります。
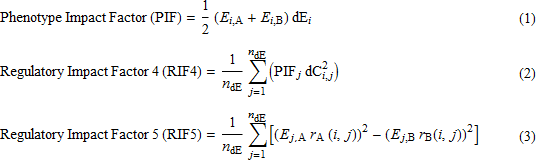
式 (1) (PIF) は2つの生物学的状態 (A と B) の間の i 番目の遺伝子の平均発現を計算し、その結果に状態 A と B の間の i 番目の遺伝子の差異的発現を掛けます。そうすることで、遺伝子の差異的発現の大きさは、全体の遺伝子の発現レベルによって重み付けされます。PIF は、次に、それぞれの差異的発現遺伝子の PIF 値と、状態 A と B の間の各差異的発現遺伝子 (上記の類推の "奴隷" 遺伝子) と各転写調整遺伝子 (上記の類推の "支配者" 遺伝子) 間の (Spearman の相関係数を用いて計算される) 差異的共発現を掛ける式 (2) (RIF4) を計算するのに使われます。それぞれの差異的発現遺伝子のこれらの計算を合計することにより、状態 A と B の間の表現型の変化を動かす最も重要な調整遺伝子の優先順位のリストを得ることができます。式 (2) はどの調整遺伝子が最も豊富な差異的発現遺伝子と一貫して高い差異的共発現になるか、という質問に対する回答を提供するように設計されています。式 (3) (RIF5) は式 (2) (RIF4) の代替のメトリックで、これもまた表現型の変化を動かす最も重要な調整遺伝子の優先順位のリストを生成しようとします。それぞれの差異的発現遺伝子の式とそれ自身と各転写調整遺伝子間の相関を2回 (状態 A で1回、状態 B で 1回) 掛けることにより、状態値の差を計算することができ、そして最も重要な調整遺伝子の代替の優先リストを得るために、各々の差異的発現遺伝子を足し合わせます。式 (3) は、どの調整遺伝子が差異的発現遺伝子の豊富さを予測する最も調整された能力を持つか?という質問に答えるように設計されています。これらの式の詳細は [3-4] で提示されていますが、RIFA でこれらのメトリックを使う背後にある基本的な考え方は簡単です。(よく考えられた実験から) 遺伝子発現のデータが RIFA に与えられたとき、アルゴリズムは (表現型の違いを生むマスター遺伝子を同定する) "音を作成するライフル銃弾" の位置を三角測量する "サウンドオフ構造のエコー" (差異的遺伝子発現データ) を使用することができます。
RIFA はテンプレート・ドリブンです。つまり、入力する情報のためのテンプレートとして使われるノートブックセルの中で、アルゴリズムはユーザー定義のいくつかの情報が与えられるのを想定していることを意味します。RIFA は AftyDGED [5] からの出力を処理するように設計されたため、リーダーが AffyDGED に精通していることが想定されます。RIFA の機能は、腫瘍肝臓組織の遺伝子発現プロファイルと、肝硬変肝臓組織を比較するマイクロアレイ研究からのデータを使って説明されます。この研究で使われ、ここで提示する全てのマイクロアレイデータは NCBI の Gene Expression Omnibus portal (www.ncbi.nlm.nih.gov/geo) で、アクセス番号 GSE17548 を使って利用できます。
Mathematica に大きなデータセットをインポートするため、利用可能なメモリを拡張するという明確な目的のために、テンプレートセルは意図的に Java の再インストールを行うコマンドから始まります。
テンプレートセルの残りの部分は、ユーザー入力を必要とするいくつかの変数を定義します。
1. timecoursedata:この変数は、RIFA によって処理を行うため、スプレッドシート形式のマイクロアレイ遺伝子発現データを含むディレクトリを指し示します。この変数はその名前の一部として "timecourse" という言葉を使用していますが、マイクロアレイデータは時間経過実験の一部である必要はありません。データのスプレッドシート形式は変更できず、厳格な形式を必要とします。指示を分かりやすくするために、この論文で記述される timecoursedata のスクリーンショットを付け加えます (図2) 。
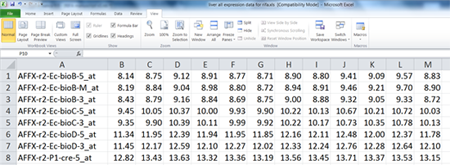
図2.変数 <timecoursedata> のための遺伝子発現データの形式
列 A は研究で使われるマイクロアレイチップから固有の転写識別情報を含みます。列 B から X は、研究の同一の実験下でのサンプル (もしくは時点) からの遺伝子発現測定を含みます。X より後の列は研究の同一の制御条件下でのサンプル (または時点) からの遺伝子発現測定を含みます。例えば、上記の肝臓研究では、複数の患者からの腫瘍サンプルはランダムに6つのグループに配置され、AffyDGED によって肝硬変肝臓組織の6つのグループと比較されました。列 B は AffyDGED で処理した腫瘍サンプルの第1グループのための遺伝子発現測定 (差異的発現でなく、転写存在量) を含み、列 C は腫瘍サンプルの第2グループからの遺伝子発現測定を含むというようになっています。この例では、腫瘍 (実験条件) データを含む最後の列は列 G です。列 H は肝硬変 (制御条件) 組織の第1のグループからの遺伝子発現測定を含む最初の列で、列Iは肝硬変組織の第2グループからというようになっています。列 B と H がそれぞれ、どのように腫瘍と肝硬変組織の最初のグループの AffyDGED の処理からの出力を含んでいるかに注目してください。
2. Conditiononecol:これは、実験条件データを含むように、timecoursedata 内の最初と最後の列の位置を定義する短いリストを含みます。
3. Conditiontwocol:これは、制御条件データを含むように、timecoursedata 内の最初と最後の列の位置を定義する短いリストを含みます。
4. rawtranscriptionreg:この変数は、マイクロアレイチップ上にある既知または疑いのある転写因子遺伝子の (1つの列に編成される) リストを含む、スプレッドシートファイルの位置を指します。ここで使用されるファイルは、転写プロセスにリンクされている遺伝子に対する Affymetrix の Human Genome U133 Plus 2.0 chip (www.affymetrix.com で利用可能) の注釈ファイルの生物学的プロセス列を解析することによって作成されました。遺伝子のこのグループを参照するプローブセット識別子は、転写因子遺伝子のリストを構築するために使用されました。
5. Rawdegenes:このリンクは、AffyDGED を使って (上記の) timesoursedata で参照される実験グループと制御グループを処理することにより作られた (プローブセット ID によって参照され、列に整理された) 差異的発現遺伝子のリストを含む、スプレッドシートファイルを指し示します。この肝臓の例には、肝硬変肝臓の6つのグループと腫瘍肝臓の6つのグループを比較するために、AffyDGED を使って作られた差異的発現遺伝子の6つの列があります。
6. affyginlocation:この変数は、RIFA からの出力に注釈をつけるために必要な情報を提供する Affymetrix の .gin (遺伝子情報) ファイルを見つけるためのディレクトリの場所を保持します。
7. savelocationroot:この変数は、ユーザーが解析の最終結果を保存したい場所を保持します。
8. studyname:この変数は、ユーザーに研究固有の情報と一緒に RIFA によって作成された出力ファイルに名前をつけることを許します。
RIFA によって行われた最初のタスクは、ダウンストリームの計算を容易にするために、読み込み、構文解析、生データの整理を含みます。
このコードの最初のセクションが完了すると、転写因子遺伝子 (上記の類推の "支配者" 遺伝子) は、それぞれのペアのスピアマンの順位相関係数の計算を容易にするために、差異的発現遺伝子 (上記の類推の "奴隷" 遺伝子) にグループ化されます。
RIFA は、スピアマンの順位相関係数を計算することによって進みます。これはそれぞれの遺伝子発現測定のベクトルが、重複したエントリの存在の検証を必要とします。また、スピアマンの rho を計算するために特別な処理を必要とします。これが下記の tieCheck モジュールの目的です。tieCheck の結果に基づいて、コードは、速度を最大にするために、必要に応じて、関数がリスト可能であることとコンパイル可能な式を使うことを利用し、スピアマンの rho の計算を最適化するために spearmanControl モジュールを呼び出します。



注意:この論文で説明されているデータを使って実行するには膨大な計算が必要で、コードの次のセグメントは完了までに (コンピュータの速度に依存しますが) おそらく 20-40 分かかり、およそ 28 GB の RAM を消費します。より少ない RAM のマシン上の計算も終了するでしょうが、かなりのハードドライブの使用が必要になり、計算が相当遅くなります。

腫瘍と肝硬変肝臓の "支配者" と "奴隷" 遺伝子の相関をペアリング (とサンプリング) することにより、満足させる (しかし生物学的に驚くことではない) パターンを明らかにしました (図3) 。腫瘍肝臓より肝硬変肝臓に、"支配者" と "奴隷" 遺伝子間の強い正の相関があることは明らかです。これは生物学者に長い間知られていることを支持します。すなわち、腫瘍組織の遺伝子調整はかなり協調に欠けます。
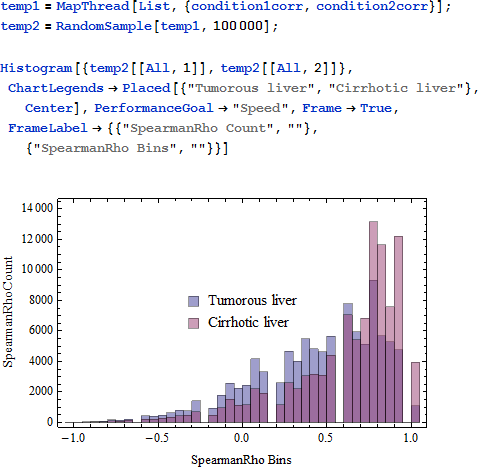
図3.腫瘍と肝硬変肝臓生検における調整遺伝子と差異的発現遺伝子間のスピアマン順位相関係数のヒストグラム
以下の相関の計算に従って、RIFA は上記の式 (1) 、(2) 、(3) の PIF、RIF4、RIF5 を含むいくつかの重要なメトリックを計算します。

PIF (式 (1)) 、RIF4 (式 (2)) 、RIF5 (式 (3)) の結果のプロット (図4) は、3つのメトリックのそれぞれの双方向性を明らかにします。言い換えると、メトリックの使用にかかわらず、-8 と +8 は基礎生物学にも同様に影響力があります。上記のメトリックの計算で使われる遺伝子発現測定が log2 スケールで表されることを思い出すと、これは理にかなっています。
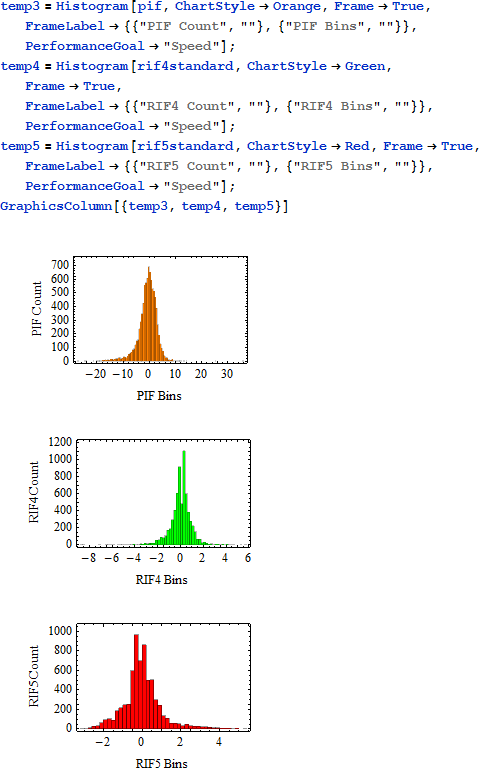
図4.RIFA アルゴリズムで使われる3つの主要なメトリックのそれぞれのヒストグラム。正および負の値は等しく重要であると解釈すべきです。(例えば、発現の中で 4倍下がった遺伝子は、発現の中で 4倍上がった遺伝子と等しく重要そうです。)
メトリックの計算が完了した後、4つのファイルが Mathematica と Microsoft Excel で直接使用可能なファイル形式で、すべての結果を含んで出力されます。ファイルの 1つのセットは、フレーズ "RifSortByAvg" が付け加えられ、RIF4 と RIF5 の値の平均によってソートされたテーブル形式の以下の情報を含みます。
ファイルの2番目のセットは、フレーズ "sortedPIF" が付け加えられ、PIF 値によってソートされたテーブル形式の以下の情報を含みます。
前述したように、"支配者" 遺伝子を同定するには RIF4 と RIF5 の結果が最も有用で、"奴隷" 遺伝子を同定するには PIF の結果が最も有用です。"支配者" と "奴隷" 遺伝子の両方とも、2つの状態間の表現型の違いを作成することに影響力を持つことができます。
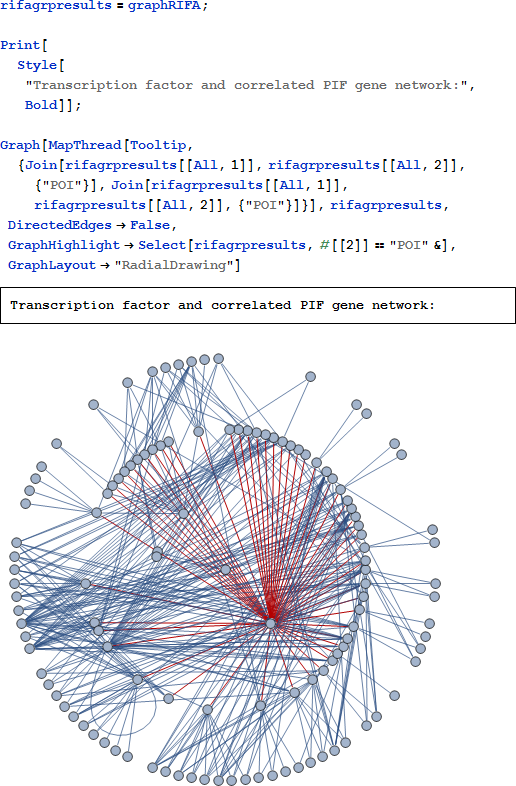
RIFA の最終的な出力は、最も強く相関し、高い影響の PIF スコアと、最も高い影響の RIF スコアを関係させたネットワークグラフです。このグラフでは、正と負の平均 RIF エントリのトップ 10 と、正と負の RIF4 エントリのトップ 10、正と負の RIF5 エントリのトップ 10 が、赤い辺で興味対象の表現型とリンクされます。言い換えると、赤い辺が表現型の変化をもたらす最も責任のある "支配者" 遺伝子を強調表示しています。グラフのノードは、ノードによって表される遺伝子を同定するためにツールチップを使用しています。関心あるノードの表現型は "POI" と略されます。表現型の変化をもたらす "支配者" 遺伝子に最も責任を持ち、また最も相関のある "奴隷" 遺伝子を強調するために青い辺が使われます。このように、グラフは、"支配者" 遺伝子の表現型の変更の命令に反応する "奴隷" 遺伝子を強調します。ここでは、正の最も高い 0.5% と負の最も低い 0.5% の PIF スコアが、転写調整遺伝子と ±0.9 のスピアマンの rho の値を共有しているのであれば、転写調整遺伝子に接続されています。


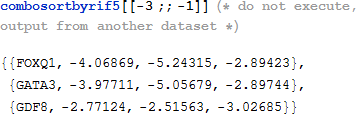
3つの主要な証拠のラインは、RIFA が仕様を設計していることを示します。証拠のライン1:表現型の変化を起こす最も直接的な責任を持つ遺伝子をハイライトするために、元は Hudson らによって記述された regulatory impact factor algorithm の Mathematica での実装を提供するように RIFA は作られました。開発中に、RIFA は和牛とピエモンテ牛間の表現型の違いを動かす最も原因となる遺伝子を強調するために、これらの著者によって使われたオリジナルのデータで調査されました。これらの品種間の最も顕著な (よく特徴付けられた) 表現型の違いは、その品種のミオスタチン (GDF8) 遺伝子の中の突然変異に起因すると知られている [7]、ピエモンテ種で増加した筋肉組織です。著者自身のデータを使用して、RIFA はその RIF5 の出力の一番下 (最も負の値 -3.02) の GDF8 を正しく識別します。
証拠のライン2:ピエモンテ/和牛のデータは、RIFA が正しいことを証明するために、完全に公開されているデータセットのみを使います。そのため、証拠のライン1は、RIFA が Hudson らの結果を再現することが可能であるため、RIFA が正常に機能しているという強い証拠を示します。たとえそうであっても、他の遺伝子発現のデータセットは分析され、RIFA の出力との比較を示す文献で議論されています。RIFA の入力に必要な、正規化された遺伝子発現データの完全なリストと、転写調整遺伝子の完全なリストは公開されていないため、RIFA の結果はこれらの他の例と同一であることはずがないことに注意してください。Reverter と同僚は、彼らの [8] からのブタの遺伝子発現データの解析を議論し、なぜその結果が間違いなく科学に知られている [9] 最も重要な性決定遺伝子の一つである遺伝子である SRY を優先しないのかを説明しようと試みました。AffyDGED と遺伝子調整遺伝子の代替リストから正規化された遺伝子発現を使った、この同じデータの再分析は、RIFA は4つ目の最も負な優先順位の遺伝子として、SRY をハイライトしないことを示します。満足できる結果ではありますが、それはまた、(RIFA を含む) 全てのアルゴリズムが、与えられた入力データの質に敏感であるという事実を強調することを示します。
証拠のライン3:上記の議論を念頭におくと、[10-11] で参照されるデータの同様の再分析は、RIFA が CDK8 [12] を 33番目の最も負の優先順位の遺伝子としてハイライトしていることを示します。このデータの Hudson の分析は、CDK8 が結腸癌の4番目に正である調整遺伝子であることをハイライトします。Hudson の解析では4番目とランク付けされているのに、なぜ RIFA は CDK8 を 33番目であるとランク付けするのでしょうか?最も合理的な解釈は、Hudson の研究では 1,292 エントリの小さい調整遺伝子のリストを利用していますが、RIFA は 6,685 の調整遺伝子を含む転写遺伝子リストを処理したことです。1,292中の4番目 (4割る 1,292) は 0.0031 で、6,685中の 33番目は 0.0049 です。転写調整遺伝子リストのサイズのパーセンテージに基づくと、RIFA の出力はアルゴリズムを開発したオリジナルの著者によって得られたものとほぼ同じになります。
提示された証拠から、RIFA が期待通りに実行されることが分かり、他の研究からの結果が、生物学的な意味をなすかどうかの確認を始めることができます。もし RIFA が正常に動作している場合、RIFA は科学的な文献中で、肝硬変および/または癌にリンクしている遺伝子をハイライトしているはずです。すべての遺伝子が肝硬変にリンクされている、もしくは癌が見つかっている、もしくは特徴付けられているわけではないことに注意してください。これは RIFA のようなプログラムを使うときに使われる値で、すなわち、遺伝子と表現型との間に新しい接続を見つけるためのものです。出力の小さなサンプルを簡単に参照するために、ここで再表示します。また、出力は、各エントリ中のデータを下記のテーブルの2つ以上の行を使うようにすることによって、印刷可能なマージン内に収まるようにフォーマットし直します。
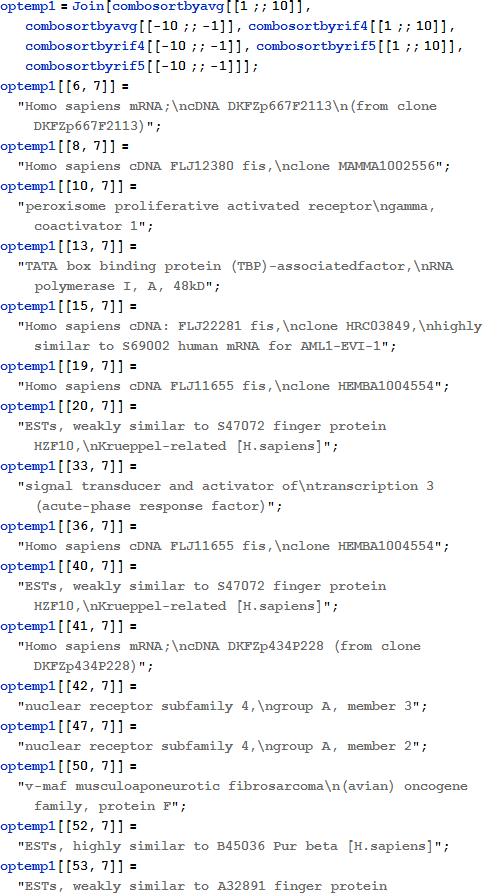
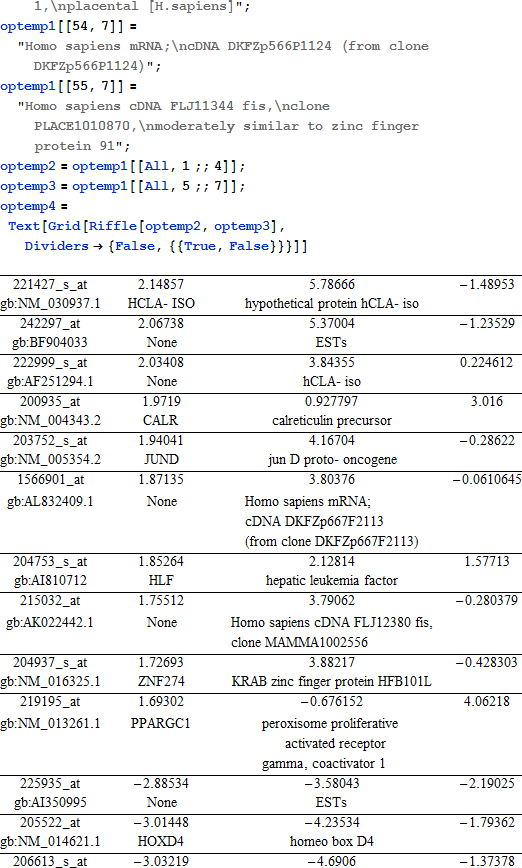
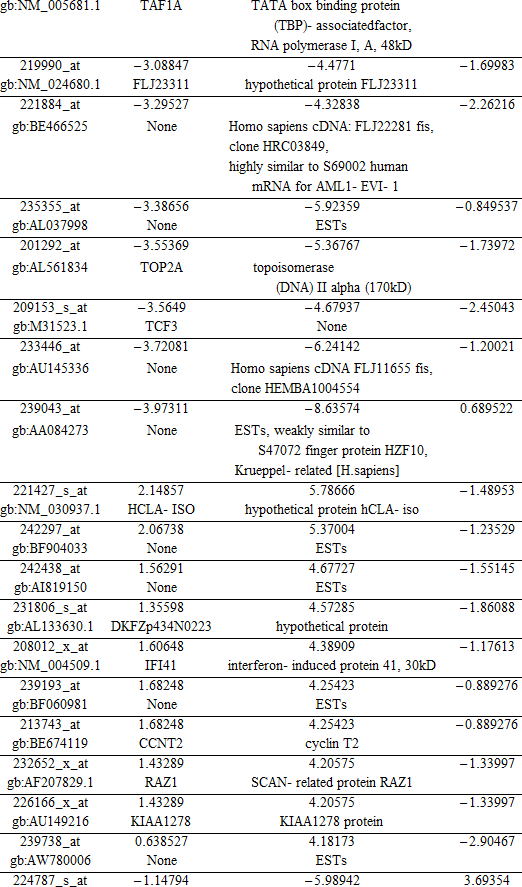
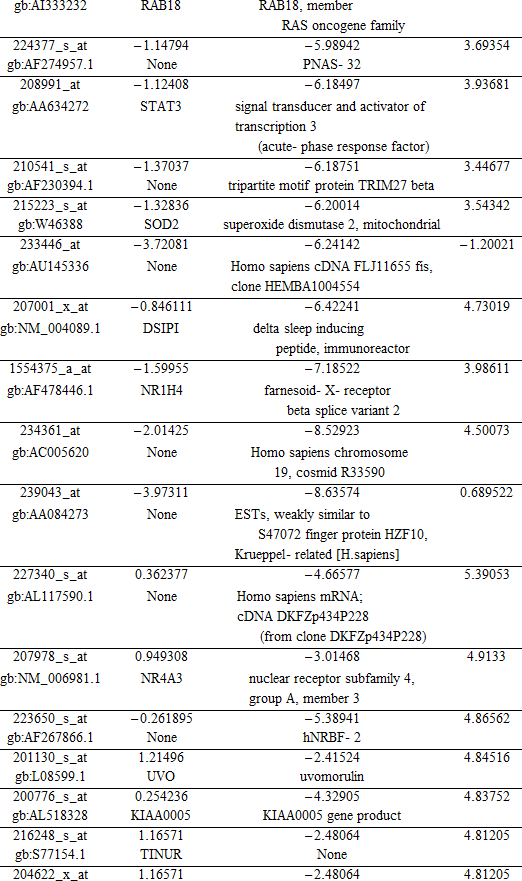
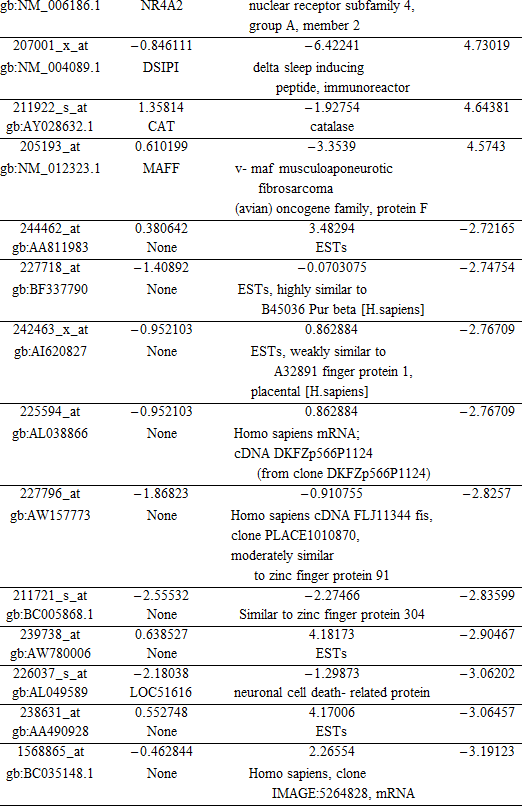
このリストでどのような情報が明らかになりますか?まず、このリストにある一意のエントリのリストを取得します (少数のエントリは RIF4 と RIF5 メトリックによって同時に優先順位を付けられるかもしれないため、複数回現れます) 。
結果のいずれかに、科学的な文献中の肝硬変もしくは腫瘍肝臓へリンクされたエントリが含まれていますか?それに関連した遺伝子名を持つエントリは、さらに研究することができる情報を持っているかもしれません。
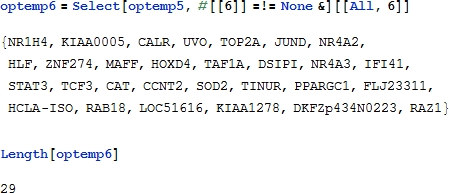
RIFA は、PubMed (www.ncbi.nlm.nih.gov/pubmed) の中で検索することができる 29個の一意の遺伝子名のリストを生成します。肝疾患検索語に関連したこれらの遺伝子の文献検索を実行することは、表1で記述される結果を生成します。
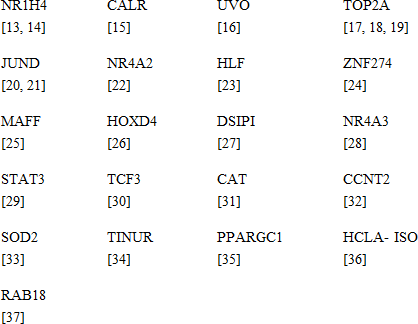
表1.次の検索語の1つもしくは複数の組み合わせにおける、上記の遺伝子名を使用した PubMed 文献検索の結果:"liver cancer"、"liver cirrhosis"、"cancer" 記載されている列挙は、一般的に発見された合計ヒット数の小さなサンプルを表します。
それらと関連した遺伝子名を持つ 29 の RIFA 出力エントリのうちの 21個は、RIFA は肝硬変と腫瘍肝組織の間で観察される表現型の変化をもたらす遺伝子を増やすことを示唆する、各遺伝子と疾患表現型検索語 "liver cirrhosis"、"liver cancer"、"canser" の間の納得できる接続を生じます。
29 遺伝子のうちの残りの 8個は、これらの疾患の表現型とリンクしている科学的な文献において証拠を示しません。これについての説明はたくさんありますが、これらの遺伝子が、実際に、疾患の表現型にリンクされているが、まだ科学界によって特徴付けられていないという可能性を排除することはできません。現時点では、これらの8個の遺伝子が疾患表現型にリンクされているか、リンクされていないかの結論を下すことは不可能です。同じ結論は、それらに関連付けられた遺伝子名を持たない他の 25個の RIFA 出力エントリにも認める必要があります。言い換えると、RIFA は、肝組織における腫瘍転移の肝硬変に関連付けられた 25個の新しい潜在的な "支配者" 遺伝子を同定しました。これらは、研究の貴重な新しい道を示すかもしれません。
RIFA の性能を評価するために、いくつかの異なるサイズと複雑さの公開されているデータセットを分析しました。表2の第1列は、NCBI の遺伝子発現オムニバスで利用可能な各データセットのシリーズ・アクセッション番号を示しています。時間は、4.48 Ghz にオーバークロックされた Intel Core i5-2500k プロセッサーを使って、Windows 7 (64 bit) 上で Mathematica 9.0.1 を実行して取得しました。システムメモリの合計のサイズは 32 GB です。すべての報告された時間は、フレッシュなカーネルを使用しています。
表2は、小さなデータセットは一分未満で簡単に処理することができることを示しています。一方、数千の転写調整遺伝子、差異的発現遺伝子、複数の時点を伴う非常に大きなデータセットは、30分以上かかることがあります。大きなデータセットを用いて測定されたかなりのメモリ消費によって証明されるように、RIFA のコードベースは、速度を上げることが可能な場合には Listable 属性を持つ関数を利用します。これはコンピュータのメモリ資源を多く消費します。
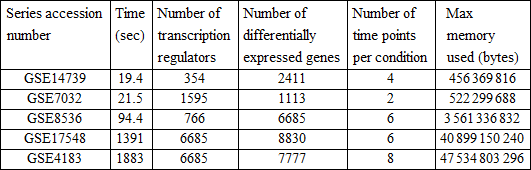
表2.公開されている異なる 5つのデータセットを使った RIFA の実行時間
遺伝子発現の変化は、疾患組織から健康な組織を見分けるものの核心です。疾患の背後にある謎の解明の一部は、疾患特色とその違いを関連付ける遺伝子発現の違いの制御に対して、最も直接的に責任がある遺伝子を同定することに集中します。RIFA の実装は Mathematica ユーザーコミュニティに、生物に存在する数千の遺伝子に理性的に優先順位をつけ、関心のある特定の特性に彼らの行動を繋げるために、生物医学研究者によって使われる説得力のあるアルゴリズムをもたらします。
| [1] | M. Schena, D. Shalon, R. W. Davis, and P. O. Brown, “Quantitative Monitoring of Gene Expression Patterns with a Complementary DNA Microarray,” Science, 270(5235), 1995 pp. 467-470. doi:10.1126/science.270.5235.467. |
| [2] | N. J. Hudson, A. Reverter, and B. P. Dalrymple, “A Differential Wiring Analysis of Expression Data Correctly Identifies the Gene Containing the Causal Mutation,” PLOS Computational Biology, 5(5): e1000382, 2009. doi:10.1371/journal.pcbi.1000382. |
| [3] | A. Reverter, N. J. Hudson, S. H. Nagaraj, M. Pérez-Enciso, and B. P. Dalrymple, “Regulatory Impact Factors: Unraveling the Transcriptional Regulation of Complex Traits from Expression Data,” Bioinformatics, 26(7), 2010 pp. 896-904. doi:10.1093/bioinformatics/btq051. |
| [4] | N. J. Hudson, B. P. Dalrymple, and A. Reverter, “Beyond Differential Expression: The Quest for Causal Mutations and Effector Molecules,” BMC Genomics, 13(356), 2012. doi:10.1186/1471-2164-13-356. |
| [5] | T. Allen, “Detecting Differential Gene Expression Using Affymetrix Microarrays,” The Mathematica Journal, 15, 2013. doi:10.3888/tmj.15-11. |
| [6] | G. Yildiz, A. Arslan-Ergul, S. Bagislar, O. Konu, H. Yuzugullu, O. Gursoy-Yuzugullu, N. Ozturk, C. Ozen, H. Ozdag, E. Erdal, S. Karademir, O. Sagol, D. Mizrak, H. Bozkaya, H. Gokhan Ilk, O. Ilk, B. Bilen, R. Cetin-Atalay, N. Akar, and M. Ozturk, “Genome-Wide Transcriptional Reorganization Associated with Senescence-to-Immortality Switch during Human Hepatocellular Carcinogenesis,” PLOS One, May 15, 2013. doi:10.1371/journal.pone.0064016. |
| [7] | C. Berry, M. Thomas, B. Langley, M. Sharma, and R. Kambadur, “Single Cysteine to Tyrosine Transition Inactivates the Growth Inhibitory Function of Piedmontese Myostatin,” American Journal of Physiology: Cell Physiology, 283(1), 2002. doi:10.1152/ajpcell.00458.2001. |
| [8] | M. Pérez-Enciso, A. L. J. Ferraz, A. Ojeda, and M. López-Béjar, “Impact of Breed and Sex on Porcine Endocrine Transcriptome: A Bayesian Biometrical Analysis,” BMC Genomics, 10(89), 2009. doi:10.1186/1471-2164-10-89. |
| [9] | D. Wilhelm, S. Palmer, and P. Koopman, “Sex Determination and Gonadal Development in Mammals,” Physiological Reviews, 87(1), 2007 pp. 1-28. doi:10.1152/physrev.00009.2006. |
| [10] | S. H. Nagaraj and A. Reverter, “A Boolean-Based Systems Biology Approach to Predict Novel Genes Associated with Cancer: Application to Colorectal Cancer,” BMC Systems Biology, 5(35), 2011. doi:10.1186/1752-0509-5-35. |
| [11] | O. Galamb, S. Spisák, F. Sipos, K. Tóth, N. Solymosi, B. Wichmann, T. Krenács, G. Valcz, Z. Tulassay, and B. Molnár, “Reversal of Gene Expression Changes in the Colorectal Normal-Adenoma Pathway by NS398 Selective COX2 Inhibitor,” British Journal of Cancer, 102, 2010 pp. 765-773. doi:10.1038/sj.bjc.6605515. |
| [12] | A. S. Adler, M. L. McCleland, T. Truong, S. Lau, Z. Modrusan, T. M. Soukup, M. Roose-Girma, E. M. Blackwood, and R. Firestein, “CDK8 Maintains Tumor Dedifferentiation and Embryonic Stem Cell Pluripotency,” Cancer Research, 72(8), 2012 pp. 2129-2139. doi:10.1158/0008-5472.CAN-11-3886. |
| [13] | G. Li, Y. Zhu, O. Tawfik, B. Kong, J. A. Williams, L. Zhan, K. M. Kassel, J. P. Luyendyk, L. Wang, and G. L. Guo, “Mechanisms of STAT3 Activation in the Liver of FXR Knockout Mice,” American Journal of Physiology: Gastrointestinal and Liver Physiology, 305(11), 2013 pp. G829-837. doi:10.1152/ajpgi.00155.2013. |
| [14] | G. Li, B. Kong, Y. Zhu, L. Zhan, J. A Williams, O. Tawfik, K. M Kassel, J. P Luyendyk, L. Wang, and G. L Guo, “Small Heterodimer Partner Overexpression Partially Protects against Liver Tumor Development in Farnesoid X Receptor Knockout Mice,” Toxicology and Applied Pharmacology,272(2), 2013 pp. 299-305. doi:10.1016/j.taap.2013.06.016. |
| [15] | K. J. Archer, V. R. Mas, K. David, D. G. Maluf, K. Bornstein, and R. A. Fisher, “Identifying Genes for Establishing a Multigenic Test for Hepatocellular Carcinoma Surveillance in Hepatitis C Virus-Positive Cirrhotic Patients,” Cancer Epidemiology, Biomarkers & Prevention, 18(11), 2009 pp. 2929-2932. doi:10.1158/1055-9965.EPI-09-0767. |
| [16] | M. Mareel, M. Bracke, and F. Van Roy, “Cancer Metastasis: Negative Regulation by an Invasion-Suppressor Complex,” Cancer Detection and Prevention, 19(5), 1995 pp. 451-464.www.ncbi.nlm.nih.gov/pubmed/7585733. |
| [17] | N. Wong, W. Yeo, W.-L. Wong, N. L.-Y. Wong, K. Y.-Y. Chan, F. K.-F. Mo, J. Koh, S. L. Chan, A. T.-C. Chan, P. B.-S. Lai, A. K.-K. Ching, J. H.-M. Tong, H.-K. Ng, P. J. Johnson, and K.-F. To, “TOP2A Overexpression in Hepatocellular Carcinoma Correlates with Early Age Onset, Shorter Patients Survival and Chemoresistance,” International Journal of Cancer, 124(3), 2009 pp. 644-652. doi:10.1002/ijc.23968. |
| [18] | N. B. Dawany, W. N. Dampier, and A. Tozeren, “Large-Scale Integration of Microarray Data Reveals Genes and Pathways Common to Multiple Cancer Types,” International Journal of Cancer, 128(12), 2011 pp. 2881-2891. doi:10.1002/ijc.25854. |
| [19] | J. M. Llovet, Y. Chen, E. Wurmbach, S. Roayaie, M. I. Fiel, M. Schwartz, S. N. Thung, G. Khitrov, W. Zhang, A. Villanueva, C. Battiston, V. Mazzaferro, J. Bruix, S. Waxman, and S. L. Friedman, “A Molecular Signature to Discriminate Dysplastic Nodules from Early Hepatocellular Carcinoma in HCV Cirrhosis,” Gastroenterology, 131(6), 2006 pp. 1758-1767.doi:10.1053/j.gastro.2006.09.014. |
| [20] | S. C. Hasenfuss, L. Bakiri, M. K. Thomsen, E. G. Williams, J. Auwerx, and E. F. Wagner, “Regulation of Steatohepatitis and PPAR Signaling by Distinct AP-1 Dimers,” Cell Metabolism,19(1), 2014 pp. 84-95. doi:10.1016/j.cmet.2013.11.018. |
| [21] | M. R. Ebrahimkhani, F. Oakley, L. B. Murphy, J. Mann, A. Moles, M. J. Perugorria, E. Ellis, A. F. Lakey, A. D. Burt, A. Douglass, M. C. Wright, S. A. White, F. Jaffré, L. Maroteaux, and D. A. Mann, “Stimulating Healthy Tissue Regeneration by Targeting the 5-HT2B Receptor in Chronic Liver Disease,” Nature Medicine, 17(12), 2011 pp. 1668-1673. doi:10.1038/nm.2490. |
| [22] | Z.-Q. Pan, Z.-Q. Fang, and W.-L. Lu, “Characteristics of Gene Expression of Adrenal Cortical Steroid Synthetase and Its Regulatory Factor in Mice with H22 Liver Cancer of Different Patterns,” Zhongguo Zhong Xi Yi Jie He Za Zhi, 31(1), 2011 pp. 85-89.www.unboundmedicine.com/medline/citation/21434351. |
| [23] | J. Dzieran, J. Fabian, T. Feng, C. Coulouarn, I. Ilkavets, A. Kyselova, K. Breuhahn, S. Dooley, and N. M. Meindl-Beinker, “Comparative Analysis of TGF-/Smad Signaling Dependent Cytostasis in Human Hepatocellular Carcinoma Cell Lines,” PLOS One, Aug 22, 2013.doi:10.1371/journal.pone.0072252. |
| [24] | J. W. Prokop, F. J. Rauscher 3rd, H. Peng, Y. Liu, F. C. Araujo, I. Watanabe, F. M. Reis, and A. Milsted, “MAS Promoter Regulation: A Role for Sry and Tyrosine Nitration of the KRAB Domain of ZNF274 as a Feedback Mechanism,” Clinical Science, 126(10), 2014 pp. 727-738.doi:10.1042/CS20130385. |
| [25] | A. Martínez-Hernández, H. Gutierrez-Malacatt, K. Carrillo-Sánchez, Y. Saldaña-Alvarez, A. Rojas-Ochoa, E. Crespo-Solis, A. Aguayo-González, A. Rosas-López, J. M. Ayala-Sanchez, X. Aquino-Ortega, L. Orozco, and E. J Cordova, “Small MAF Genes Variants and Chronic Myeloid Leukemia,” European Journal of Haematology, 92(1), 2014 pp. 35-41.doi:10.1111/ejh.12211. |
| [26] | J. Shen, S. Wang, Y.-J. Zhang, M. A. Kappil, H. C. Wu, M. G. Kibriya, Q. Wang, F. Jasmine, H. Ahsan, P.-H. Lee, M.-W. Yu, C.-J. Chen, and R. M. Santella, “Genome-wide Aberrant DNA Methylation of MicroRNA Host Genes in Hepatocellular Carcinoma,” Epigenetics, 7(11), 2012 pp. 1230-1237. doi:10.4161/epi.22140. |
| [27] | C. Paulin and Y. Chamay, “Demonstration of Delta Sleep Inducing Peptide in a Strain of Human Small Cell Lung Cancer by Immunocytology,” Comptes Rendus de L’Academie des Sciences. Serie III, Sciences de la vie, 314(6), 1992 pp. 259-262.www.ncbi.nlm.nih.gov/pubmed/1318772. |
| [28] | U. Flucke, B. B. J. Tops, M. A. J. Verdijk, P. J. H. van Cleef, P. H. van Zwam, P. J. Slootweg, J. V. M. G. Bovée, R. G. Riedl, D. H. Creytens, A. J. H. Suurmeijer, and T. Mentzel, “NR4A3 Rearrangement Reliably Distinguishes between the Clinicopathologically Overlapping Entities Myoepithelial Carcinoma of Soft Tissue and Cellular Extraskeletal Myxoid Chondrosarcoma,”Virchows Archiv, 460(6), 2012 pp. 621-628. doi:10.1007/s00428-012-1240-0. |
| [29] | G. Ramakrishna, A. Rastogi, N. Trehanpati, B. Sen, R. Khosla, and S. K. Sarin, “From Cirrhosis to Hepatocellular Carcinoma: New Molecular Insights on Inflammation and Cellular Senescence,” Liver Cancer, 2(3-4), 2013 pp. 367-383. doi:10.1159/000343852. |
| [30] | M. Slyper, A. Shahar, A. Bar-Ziv, R. Z. Granit, T. Hamburger, B. Maly, T. Peretz, and I. Ben-Porath, “Control of Breast Cancer Growth and Initiation by the Stem Cell-Associated Transcription Factor TCF3,” Cancer Research, 72(21), 2012 pp. 5613-5624.doi:10.1158/0008-5472.CAN-12-0119. |
| [31] | Q. Wang, W. Chen, L. Bai, W. Chen, M. T. Padilla, A. S. Lin, S. Shi, X. Wang, and Y. Lin, “Receptor-Interacting Protein 1 Increases Chemoresistance by Maintaining Inhibitor of Apoptosis Protein Levels and Reducing Reactive Oxygen Species through a MicroRNA-146a-Mediated Catalase Pathway,” Journal of Biological Chemistry, 289(9), 2014 pp. 5654-5663.doi:10.1074/jbc.M113.526152. |
| [32] | C. Simone and A. Giordano, “Abrogation of Signal-Dependent Activation of the cdk9/cyclin T2a Complex in Human RD Rhabdomyosarcoma Cells,” Cell Death & Differentiation, 14(1), 2007 pp. 192-195. doi:10.1038/sj.cdd.4402008. |
| [33] | P. Nahon, A. Sutton, P. Rufat, M. Ziol, H. Akouche, C. Laguillier, N. Charnaux, N. Ganne-Carrié, V. Grando-Lemaire, G. N’Kontchou, J.-C. Trinchet, L. Gattegno, D. Pessayre, and M. Beaugrand, “Myeloperoxidase and Superoxide Dismutase 2 Polymorphisms Comodulate the Risk of Hepatocellular Carcinoma and Death in Alcoholic Cirrhosis,” Hepatology, 50(5), 2009 pp. 1484-1493. doi:10.1002/hep.23187. |
| [34] | Y. Han, H. Cai, L. Ma, Y. Ding, X. Tan, Y. Liu, T. Su, Y. Yu, W. Chang, H. Zhang, C. Fu, and G. Cao, “Nuclear Orphan Receptor NR4A2 Confers Chemoresistance and Predicts Unfavorable Prognosis of Colorectal Carcinoma Patients Who Received Postoperative Chemotherapy,”European Journal of Cancer, 49(16), 2013 pp. 3420-3430. doi:10.1016/j.ejca.2013.06.001. |
| [35] | B. Wang, S.-H. Hsu, W. Frankel, K. Ghoshal, and S. T. Jacob, “Stat3-Mediated Activation of MicroRNA-23a Suppresses Gluconeogenesis in Hepatocellular Carcinoma by Down-Regulating Glucose-6-Phosphatase and Peroxisome Proliferator-Activated Receptor Gamma, Coactivator 1 Alpha,” Hepatology, 56(1), 2012 pp. 186-197. doi:10.1002/hep.25632. |
| [36] | H. Shimada, K. Nakashima, T. Ochiai, Y. Nabeya, M. Takiguchi, F. Nomura, and T. Hiwasa, “Serological Identification of Tumor Antigens of Esophageal Squamous Cell Carcinoma,”International Journal of Oncology, 26(1), 2005 pp. 77-86. doi:10.3892/ijo.26.1.77. |
| [37] | X. You, F. Liu, T. Zhang, Y. Li, L. Ye, and X. Zhang, “Hepatitis B Virus X Protein Upregulates Oncogene Rab18 to Result in the Dysregulation of Lipogenesis and Proliferation of Hepatoma Cells,” Carcinogenesis, 34(7), 2013 pp. 1644-1652. doi:10.1093/carcin/bgt089. |
| T. D. Allen, “RIFA: A Differential Gene Connectivity Algorithm,” The Mathematica Journal, 2015. dx.doi.org/doi:10.3888/tmj.17-2. | |
追加の電子ファイル:
Todd Allen は、クリ胴枯病病原体とクリ胴枯病菌の遺伝子発現の変化の研究のために、カスタム cDNA マイクロアレイチップを開発したメリーランド大学でのポストドクター時代に、Mathematica を使って計算生物学を行うことに興味を待ちました。