 |
| サイトマップ | |
|
 |
| サイトマップ | |
|
 |
 |
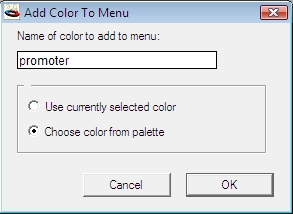 |
origin of replication" を入力し、OK ボタンを押します。色見本の中から色を選択して下さい。これで、「複製起点色」の設定が終わりました。Format|Color メニューコマンドを選択すると、この色が登録されています。同様にして、DNA のプロモーターやエンハンサー、その他のサイトを簡単に区別することができます。このようにユーザー定義した色を使ってコンストラクトを表現すると、一目でその特徴をつかむことができます。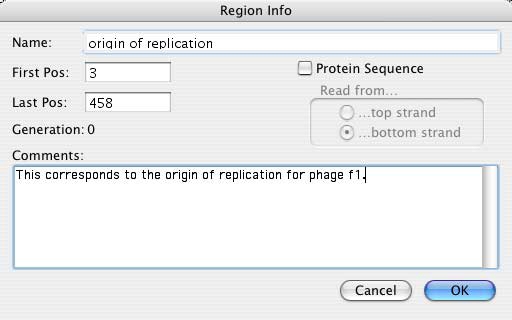 |
 |