チュートリアル 16:PCR 解析
PCR 解析コード(※)を利用して、ショウジョウバエ(Drosophila)の hsp70 遺伝子のプロモーター領域を増幅してみましょう。
※GCK uses Primer3 PCR code developed at the Whitehead Institute for Biomedical
Research and used with permission. This code is Copyright (c) 1996, 1997, 1998, 1999,
2000, 2001, 2004 Whitehead Institute for Biomedical Research. All rights reserved. The
algorithm for this software is published: Steve Rozen and Helen J. Skaletsky (2000)
Primer3 on the WWW for general users and for biologist programmers. In: Krawetz S, Misener S (eds) Bioinformatics Methods and Protocols: Methods in Molecular Biology.
Humana Press, Totowa, NJ, pp 365-386.
- Tutorials フォルダにある Hsp70 タンパク質ファイルを開きます。
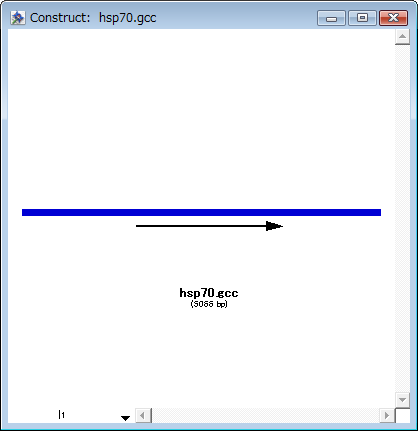
- DNA の下にある矢印は、このショウジョウバエの熱ショック遺伝子 (heat
shock gene) に使われる実際の転写産物 (transcript) です。この転写産物の上流 300 nts の領域を選択して増幅することにしましょう。この部分に重要なプロモーター要素があると考えられるからです。矢印をダブルクリックしてそれに対応する DNA 断片を選択してください。コンストラクトウィンドウの左下には選択したヌクレオチド領域
1617-3677 が表示されます。
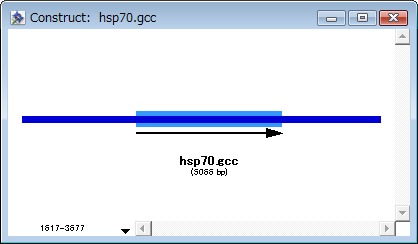
- ウィンドウ表示をシークエンス・ビューに切り替えてください(Construct > Display > Display Sequence)。同一の領域が選択されているはずです。
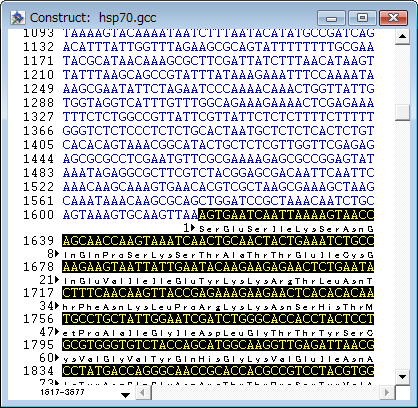
シークエンス番号 1616 の直前にある最初のヌクレオチドをマウスでクリックし、「上流」に向かってマウスをドラッグしてください。シークエンス番号 1317 付近までドラッグしたら、マウスのボタンを離します。
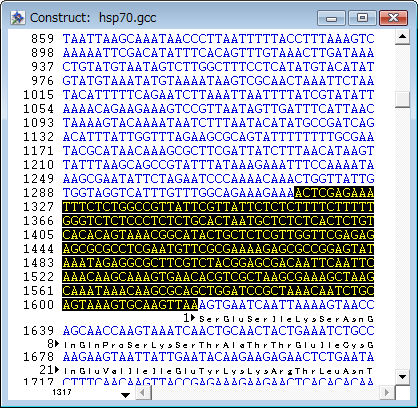
シークエンスの開始点をシフトキーを押しながら選択すれば、選択範囲を正確に指定できます。ヌクレオチド領域が 1317-1616 になるまで操作を繰り返してみてください。領域を選択できたら、そのままの状態で、ウィンドウ表示をグラフィクス・ビューに切り替えます (Construct > Display > Display Graphics または Ctrl+/ )。
- Tools メニューの PCR Analysis を選択して、PCR Analysis ダイアログを開いてください。
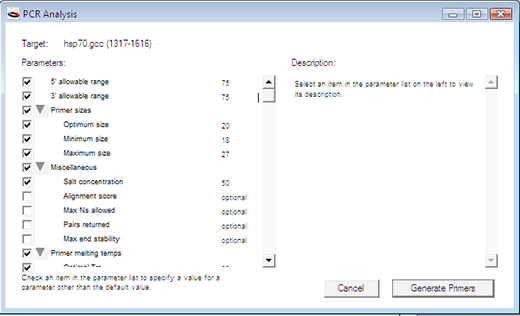
以下のようなダイアログが開きます。Parameters リストにある項目のいずれかをクリックすると、ウィンドウの左側に選択した項目の詳細内容が表示されます。設定可能なパラメータにはどのようなものがあるか簡単に目を通してください。このうち「optional」と表示されているパラメータは、数値を入力しても、しなくても構いません。ここでは、5' と 3' の許容範囲をそれぞれ 75 に設定したら、Generate
Primers ボタンを押します。
- Generate
Primers ボタンを押すと、以下のダイアログがあらわれます。元のパラメータで指定した条件に合うプライマー候補が5つ見つかりました。ウィンドウの下側には、リスト作成に際して、候補となるプライマー対がほかにどれだけあったかを示す統計データが表示されます。
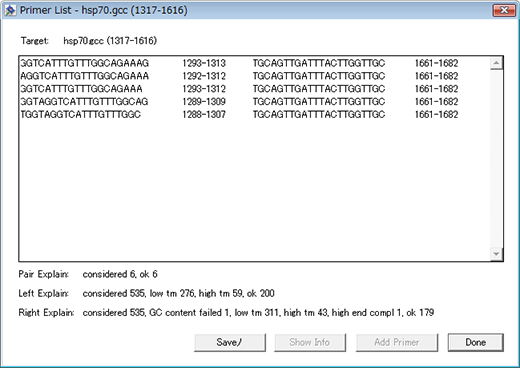
- リスト中央のプライマー対 (1293-1312, 1762-1781) を選択し、Add Primer ボタンを押してください。コンストラクトには複数のプライマーを追加することもできますが、ここで追加するのはこれだけにしておきます。プライマーを追加したら、Done ボタンを押してください。
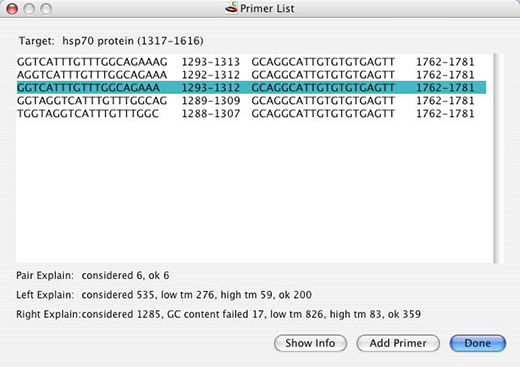
- 図のように 2つのプライマーが表示されているはずです。
プライマーのいずれかをクリックすると、プライマー対を選択できます。プライマー対をクリックして、Command-I (Mac) または Control-I (Windows) を押すと、その情報を確認することができます。Construct メニューの Get Info を選択しても同様に確認できます。
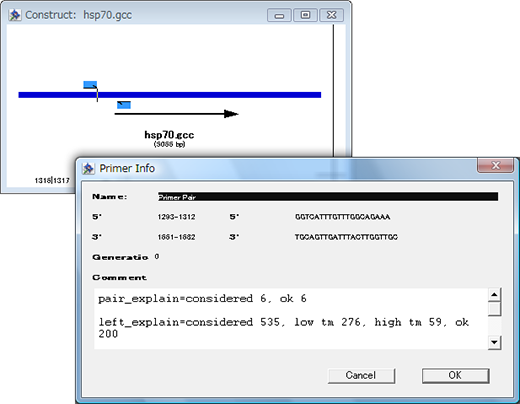
下図は、Get Info ウィンドウを表示させた例です。Get Info ウィンドウには、オリジナルのシークエンスから実際にどれだけのプライマーが作成されたのかを示す詳細な情報が表示されます。
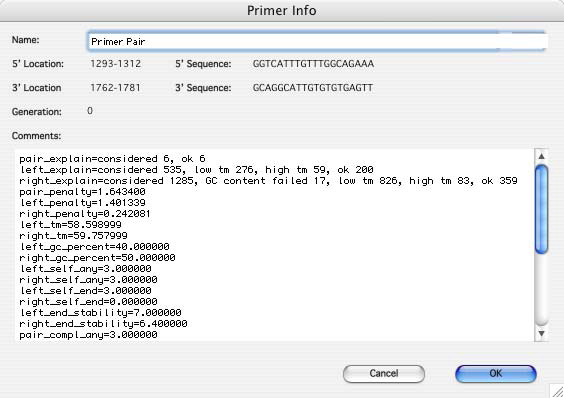
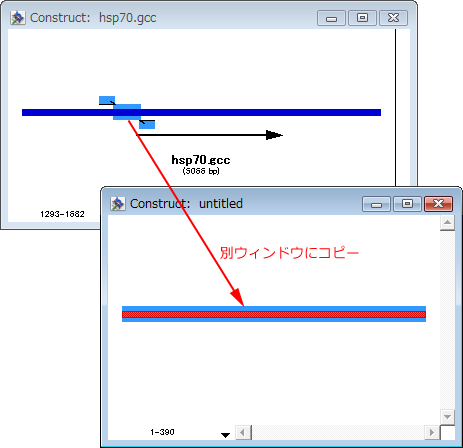
- プライマー対(プライマーのいずれでも可能)をダブルクリックすると、プライマー対によって増幅された DNA を選択できます。選択したシークエンスを別ウィンドウ(又は別アプリケーション)にコピーすれば、これを使って更に詳しく分析を進めることができます。