チュートリアル 14:GenBank を使った検索とシーケンスファイルの取り込み
次に紹介するステップは、GenBank から塩基配列を検索し、ファイルを取得するまでの手順です。取得したファイルは自動的に GCK にインポートされます。
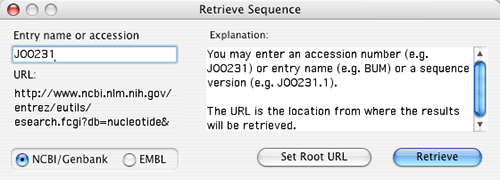
- 以下の手順を進めるには、インターネット接続環境が必要になります。接続がまだであれば、今すぐ接続してください。Tools > Deluxe Import > Retrieve Sequence を選択してプロセスを開始ます。図 2.70 のようなウィンドウが表示されます。左上の入力ボックスに ”J00231” と入力します (これ以外の accession number や塩基配列名でも構いません) 。このボックスにアクセッション番号や塩基配列名を入力したら、左下のボックスに URL が構成されるのを確認できます (GCK が実際に何を行っているかが確認できますが、直接編集することはできません。)。ここで構成された URL が目的のサーバーに送信されることになります。デフォルトで用意されたサーバーは、GenBank と EMBL ですが、Set Root URL… ボタンをクリックすることで、ローカルサーバーを指定することも可能です。
図 2.70:
Retrieve Sequence ダイアログ
 |
- Retrieve ボタンをクリックすると、ターゲット指定したデータベースに接続し、塩基配列データが取り込まれます。塩基配列データが取り込まれると、図 2.67 に示すのと同じウィンドウが表示されるはずです。ここから、前のチュートリアル (DNA セグメントのクローニングとサイレント突然変異) で紹介した同じ手順を経て、塩基配列を GCK にインポートすることができます。
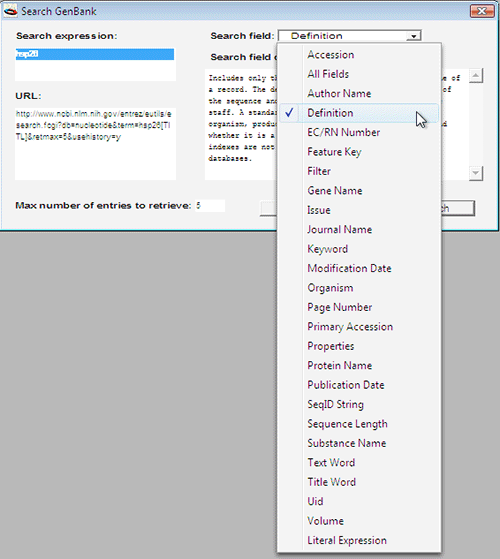
- GCK を使って GenBank を検索することもできます。File > Deluxe Import > Search GenBank を選択すると、GenBank の検索を定義する検索用ダイアログが図 2.71 のように表示されます。クエリの検索対象とするデータベースフィールドをポップアップメニューを使って定義できる点に注目してください。Search expression に検索したいキーワードを入力したら、Search ボタンをクリックします。プログレスダイアログで経過が表示されたあと、クエリーと一致する項目が一覧表示されます。図 2.68 のようなリストになっているはずです。関心のある塩基配列はすでにハードディスク内にあり、これらを GCK に直接インポートすることができます。
図 2.71:
GenBank の検索オプション
 |
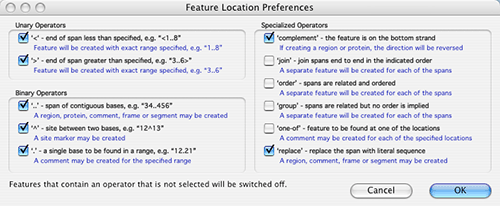
- DNA ソースファイルの features テーブルにあいまいなデータがある場合は、Location Prefs… という名前のボタンが表示されているはずです。このボタンを押すと、図 2.72 のようなウィンドウが表示され、feature locations をどのように解釈するかを定義することができます。このウィンドウには、NCBI で定義された様々な feature を GenBank ファイルで利用できるようにする手段が一覧表示されます。この中の一部は GCK の features に直接翻訳することはできませんが、そのような事態に対処できるように、このウィンドウを使って定義することができます。
図 2.72:
Location Preferences ダイアログ
 |
Deluxe Import 機能の色々な操作を実際にお試しください。Entrez を使って NCBI ウェブサイトで検索するのと同様に検索することができます。




